서 언
재료 및 방법
식물재료
복숭아 DNA 분리
FT-IR 스펙트럼과 데이터 전처리 및 다변량 통계분석
FT-IR 스펙트럼 데이터를 이용한 Classification, prediction 그리고 cross-validation
결과 및 고찰
복숭아의 FT-IR 스펙트럼 비교 분석
FT-IR 스펙트럼 다변량 통계분석 및 대사체 수준 유연관계 비교
복숭아 품종간 Classification, prediction 그리고 cross-validation
서 언
장미과(Rosaceae), 자두나무속(Prunus)에 속하는 복숭아(Prunus persica (L.) Batsch)는 낙엽성 온대과수로 중국이 원산지인 세계적인 과일이다(Ahmad et al., 2004). 우리나라에서는 사과와 감귤 다음으로 재배면적이 넓은 과종이다. 복숭아의 대표적인 형태는 과일 표면에 솜털(pubescence)의 유무에 따라 복숭아(유모종)와 천도(무모종)로 분류된다. 복숭아는 실생선발과 변이선발이 쉽고, 영양번식으로 쉽게 증식이 되기 때문에 품종 식별을 위한 분자표지 개발과 유연관계 분석이 필요하다(Hong et al., 2013).
장미과 작물 중 복숭아의 genome size는 226.6Mb로 가장 작다(Verde et al., 2013). 복숭아에 대한 유전적 다양성 및 유연관계를 파악하기 위하여 분자생물학적 분석법인 randomly amplified polymorphic DNA (RAPD), amplified fragment length polymorphism (AFLP), simple sequence repeats, sequence-related amplified polymorphism (SRAP)와 같은 다양한 DNA 마커들이 이용되고 있다(Warburton and Bliss, 1996; Casas et al., 1999; Ahmad et al., 2004; Hu et al., 2005; Xu et al., 2006; Yoon et al., 2006; Bouhadida et al., 2009; Cheng and Huang, 2009). 복숭아 품종의 친자관계를 확인하기 위해 simple sequence repeat (SSR) 마커를 이용하여 분석하였고(Yamamoto et al., 2003), 그리고 sequence characterized amplified region (SCAR) 마커를 이용하여 복숭아 품종 판별 연구가 진행되었다(Han et al., 2010). 이러한 방법들은 정확한 유전적 분석을 통해 품종에 대한 정밀한 분석이 가능하지만, 분석에 대한 복잡성 그리고 많은 시간과 비용이 소요되는 한계점을 가지고 있다. 이러한 한계점을 FT-IR fingerprinting 기술을 이용하여 사전 식별 분석을 통해 효과적으로 보완 가능하다. FT-IR spectroscopy는 화학, 농업, 제약, 식품 산업 등 다양한 연구 분야에서 널리 사용되고 있고, FT-IR 스펙트럼은 DNA의 특성을 나타낸다. 각각의 영역에서 나타내고 있는 스펙트럼은 화학적 작용기의 진동을 나타내고, DNA의 전체 화학 구조와 서로 다른 작용기작 또는 화학적 구조가 스펙트럼에 반영된다. 또한, cell의 전체 화학구조에 대한 lipids, proteins, nucleic acids 그리고 polysaccharides을 포함하고 있는 전체 화학구조를 매우 구체적으로 나타낸다(Lee and Chapman, 1986; Naumann et al., 1991; Mungur et al., 2005; Lei et al., 2011). IR 스펙트럼의 변화는 nucleic acid의 분자 모양에 의해 결정되며, 광범위한 수소 결합과 대칭구조의 phosphate 증가, 그리고 단백질의 C-OH 결합 감소를 나타낸다(Dukor, 2002). FT-IR을 이용한 분석은 미생물, 곰팡이 그리고 박테리아 식별에 사용되었고(Ngo-Thi et al., 2003; Naumann et al., 2005; Fischer et al., 2006; Preisner et al., 2007; Sundaram et al., 2012), 그리고 생식 세포질의 대사 평가(Yesudas et al., 2013), DNA를 이용한 질병과 암 식별에 사용되었다(Malins et al., 1997, 2003, 2005). 또한 바이오 마커로 이용하여 암 진단에 활용되었고(Argov et al., 2002; Sahu et al., 2008), 유전자 변형 DNA 판별에 적용 되었으며(Emura et al., 2006), indica와 japonica의 DNA 수준에서 유전적 차이가 높음을 확인하였다(Emura et al., 2006). 또한, FT-IR을 이용하여 국내에서 수집된 야생종 복숭아의 유연관계에 대해 분석된 바 있으나(Song et al., 2020), 육종내력이 있는 품종들 즉, 양친의 정보가 알려져 있거나 기존 품종의 돌연변이로 탄생한 계통들에 대한 분석은 미미하다.
복숭아 ‘유명(Yumyeong)’은 1977년 국내에서 육성된 품종으로 에틸렌이 합성되지 않아 단단한 과육이 장기간 유지되는 경육종(stony hard) 이다(Haji et al., 2001). 복숭아 품종군에서 경육종이 차지하는 비율은 매우 낮기 때문에 ‘유명’은 경육종 연구에서 자주 다루어지고 있으며 (Pan et al., 2015), 육종적으로는 저장성이 우수한 품종 육성을 위해 모본으로 활용되고 있다 (Jun et al., 2013). 현재까지 ‘유명’에서 다수의 품종들이 선발되었으나, 이들 품종 사이의 유연관계나 유전적 특성에 대한 연구는 부족한 실정이다. 따라서 본 연구는 복숭아 ‘유명(Yumyeong)’에서 파생된 인공교배품종, 자연교잡실생, 그리고 돌연변이 품종을 대상으로 genomic DNA의 FT-IR 스펙트럼 데이터를 이용하여 근연관계를 분석하고, 다양한 유전자 분석의 사전 분석 기법의 가능성을 확인하고자 하였다.
재료 및 방법
식물재료
본 연구에서는 복숭아 ‘유명’(Yumyeong, UM)과 유명(UM)을 모본으로 사용하여 육성된 4개의 육성품종 ‘유미’(Yumi, YM), ‘수미’(Soomee, SM), ‘미홍’(Mihong, MH) 그리고 ‘미스홍’(Misshong, MS), 부본으로 사용된 ‘치요마루’(Chiyomaru, JU), 유명의 자연교잡실생인 ‘유명실생-음성(Eumseong, YS)’과 ‘대월(Daewol, DW)’, 유명의 돌연변이 품종인 ‘월미(Wolmi, WM)’를 분석하였다(Table 1). 국립원예특작과학원 과수과 시험포장에 재식된 시험수를 품종별로 3주씩 3반복으로 잎 시료를 채취하였다. 각각의 잎 시료는 DNA 추출을 위해 동결건조하고 막자사발을 이용하여 분말 형태로 분쇄하였다. 분쇄된 잎 시료는 –70°C 초저온냉동고에 보관하여 분석에 사용하였다.
Table 1.
Accession, origin, pubescence of fruit skin, flesh color, and pedigree characteristics of peach varieties released by the National Institute of Horticultural and Herbal Science
복숭아 DNA 분리
수집된 식물 잎 시료를 genomic DNA 추출에 사용하였다. 동결 건조된 잎 시료 10mg을 사용하여 DNeasy Plant Mini Kit (QIAGEN, Dusseldorf, Germany) 방법에 따라 DNA를 추출하였다. 추출된 DNA는 AE buffer 를 이용하여 희석하였다. DNA 농도와 순도는 NanoDrop 2000을 이용하여 280, 260와 230nm에서 측정하였고, 추출된 DNA 순도를 확인하기 위해서 흡광비(OD260/OD280)를 계산하였다. 이 비율은 polyphenol, carbohydrate contamination 과 함께 protein을 측정하였다(Sambrook et al., 1989). AE buffer를 이용하여 DNA 최종 농도를 20ng/㎕으로 조정하였다. 또한, 효과적인 FT-IR 스펙트럼 측정을 위해 genomic DNA를 체크하여 DNA 오염도를 최소화하고, Genomic DNA 농도는 아가로스 겔 전기영동을 통해 확인하였다. 확인된 genomic DNA는 –20°C에 보관하여 실험에 사용하였다(Song et al., 2014).
FT-IR 스펙트럼과 데이터 전처리 및 다변량 통계분석
FT-IR (Fourier transform infrared) 스펙트럼 조사는 Tensor 27 (Bruker Optics GmbH, Ettlingen, Germany)과 DTGS (Deuterated triglycine sulfate) 검출기를 사용하였다(Song et al., 2014). 추출된 각각의 시료는 5µL씩 5반복으로 384-well ZnSe plate에 분주하여, 37°C hot plate에서 약 20–30분간 건조한 후 HTS-XT (Bruker Optics GmbH) 고효율 자동화 장치를 이용하여 스펙트럼을 측정하였다. 스펙트럼은 4000–400cm-1 범위에서 4cm-1 간격으로 128회 반복 측정되었고, 측정된 스펙트럼의 평균 스펙트럼을 분석에 사용하였다. Bruker 에서 제공하는 OPUS Lab (ver. 7.0, Bruker Optics Inc.)를 사용하여 FT-IR 스펙트럼 조사 및 데이터 변환을 하였고, baseline 교정을 위해 FT-IR 스펙트럼 분석 영역의 양 끝점(800–1,800cm-1)의 흡광도를 0으로 조정하였다. 실험상의 오차를 최소화하기 위하여 각 스펙트럼을 동일 면적으로 normalization을 실시하였으며, mean centering 과정을 거쳐 2차 미분하여 스펙트럼의 전처리 과정을 실시하였다.
가공된 FT-IR 스펙트럼 데이터는 NIPALS 알고리즘(Wold, 1966)을 이용하여 PCA (Principal component analysis)와 PLS-DA (Partial least squares discriminant)분석을 R 프로그램(version 3.1.2)에서 수행하였다(Fiehn et al., 2000; Trygg et al., 2007). PCA 및 PLS-DA 분석을 통해 얻어진 score를 이용하여 HCA (hierarchical clustering analysis) 분석하였으며 유사도 지수로 UPGMA (unweighted pair group method with arithmetic mean analysis)를 사용한 Euclidean distance를 측정하여 각 시료의 유연관계를 덴드로그램으로 나타냈다.
FT-IR 스펙트럼 데이터를 이용한 Classification, prediction 그리고 cross-validation
FT-IR 스펙트럼 데이터를 기반으로 DNA를 통한 품종간 예측을 위해서 PLS regression 분석을 적용하여 확립하였다. 품종간 예측을 위해 2개의 변수 X와 Y를 나누고 X 변수에는 FT-IR 스펙트럼 데이터를 Y 변수에는 품종간 DNA의 번호를 그룹화 시키고 각각의 정보를 연결시켜 PLS regression을 실시하였다. 예측 모델의 예측 능력을 테스트하기 위해 트레이닝 세트를 만들어 모델 구축에 사용하였고, 트레이닝 세트는 테스트 세트에 사용되지 않았다. X 변수와 Y 변수간의 측정 모델을 만들기 위하여 상관관계를 예측한 후 회귀계수를 사용하여 상관관계를 계산하였다. 전체의 데이터는 임의의 두 부분으로 나눴고, 먼저 트레이닝 세트를 선발 후 트레이닝 세트에 사용되지 않은 데이터를 테스트 세트로 사용하여 예측 모델을 구축하는데 사용되었다. PLS 모델의 예측 능력은 실측 값과 PLS 예측 값 사이의 정확도를 10 bootstrap 테스트하여 나타냈고, RMSEP (Root Mean Square Error of Prediction)와 MSEP (Mean Square Error of Prediction)를 측정하여 예측의 정확도를 확인하였다(Liu et al., 2006; Terhoeven-Urselmans et al., 2010).
결과 및 고찰
복숭아의 FT-IR 스펙트럼 비교 분석
복숭아 genomic DNA의 FT-IR 스펙트럼 데이터로부터 다변량통계분석을 통해 복숭아 품종간의 유연관계를 분석하였다. 각각의 복숭아 품종들은 FT-IR 스펙트럼 1,750–1,600, 1,550–1,400, 1,150–950cm-1 파장에서 특징적인 스펙트럼 패턴 변화가 나타났다(Fig. 1). FT-IR 스펙트럼 패턴의 차이는 DNA의 구조적 특성과 상호작용을 나타내는 것으로 보고되고 있다(Falk et al., 1963; Liquier et al., 1991; Zhou-Sun et al., 1997; Brewer et al., 2002; Banyay et al., 2003; Gonzalez-Ruiz et al., 2011). 1,750–1,600cm-1 부위는 전체적인 DNA의 염기구조인 C = O, C = N 그리고 C = C 형태와 –NH2결합 형태를 나타낸다(Brewer et al., 2002; Banyay et al., 2003). purine과 pyrimidine ring 구조는 1,550–1,400cm-1 부위에서 나타나며(Falk et al., 1963; Liquier et al., 1991; Zhou-Sun et al., 1997), phosphodiester-deoxyribose backbone내의 PO2- 그룹의 대칭적 구조와 비대칭적 구조는 1,100–950cm-1 부위에서 나타난다(Falk et al., 1963; Zhou-Sun et al., 1997). 이 세가지 스펙트럼 영역은 backbone과 sugar pucker 사이의 base pairing, base stacking 그리고 glycosidic bond 회전과 밀접하게 연관되어 있다(Brewer et al., 2002; Gonzalez-Ruiz et al., 2011). 이처럼 FT-IR 스펙트럼의 차이가 복숭아 품종 고유의 DNA 염기구조가 서로 다름을 의미하며, FT-IR 스펙트럼 분석을 통해 복숭아 품종별 DNA 구조적 차이와 이로 인한 유전적 차이를 확인할 수 있을 것으로 판단된다.
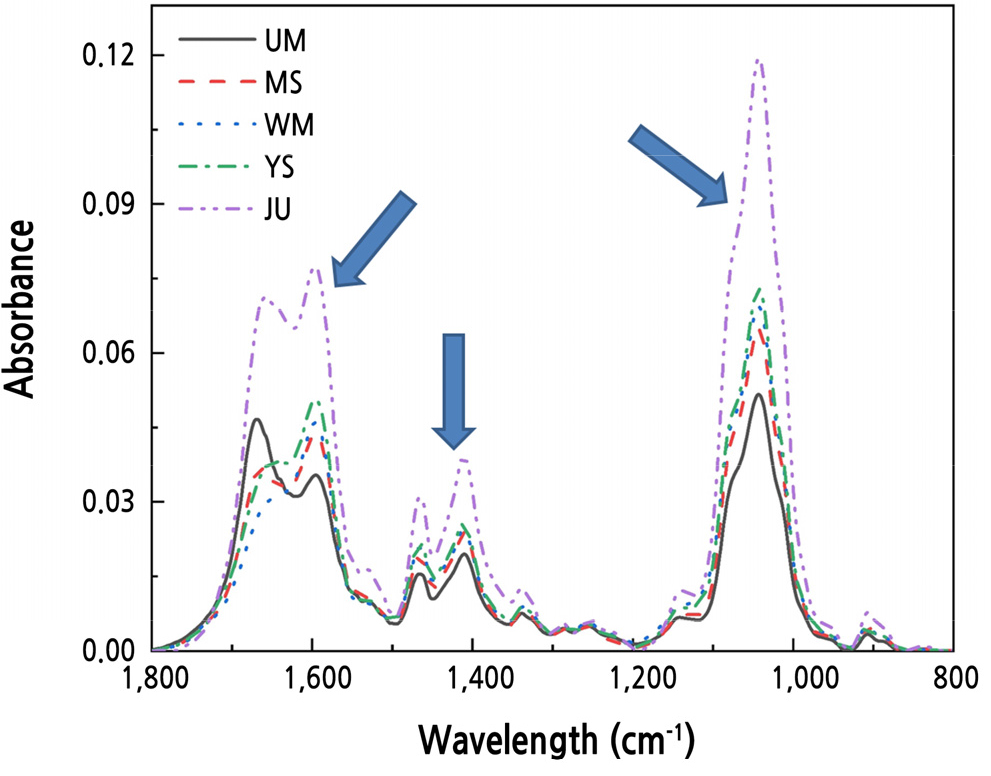
Fig. 1.
Representative FT-IR spectral from genomic DNA of peach leaves. FT-IR spectral ranges showed quantitative information of 1,750–1,600 cm-1 (amide I (N-H stretching) and amide II (C = O stretching vibrations)), 1,550–1,400 cm-1 (purine and pyrimidine ring), and 110–950 cm-1 (PO2- ionized asymmetric and symmetric stretching). UM : Yumyeong, MS : Misshong, WM : Yumyeong Mutation (Wolmi), YS : Yumyeong Seedling (Eumseong), JU : Chiyomaru. *n = 5.
FT-IR 스펙트럼 다변량 통계분석 및 대사체 수준 유연관계 비교
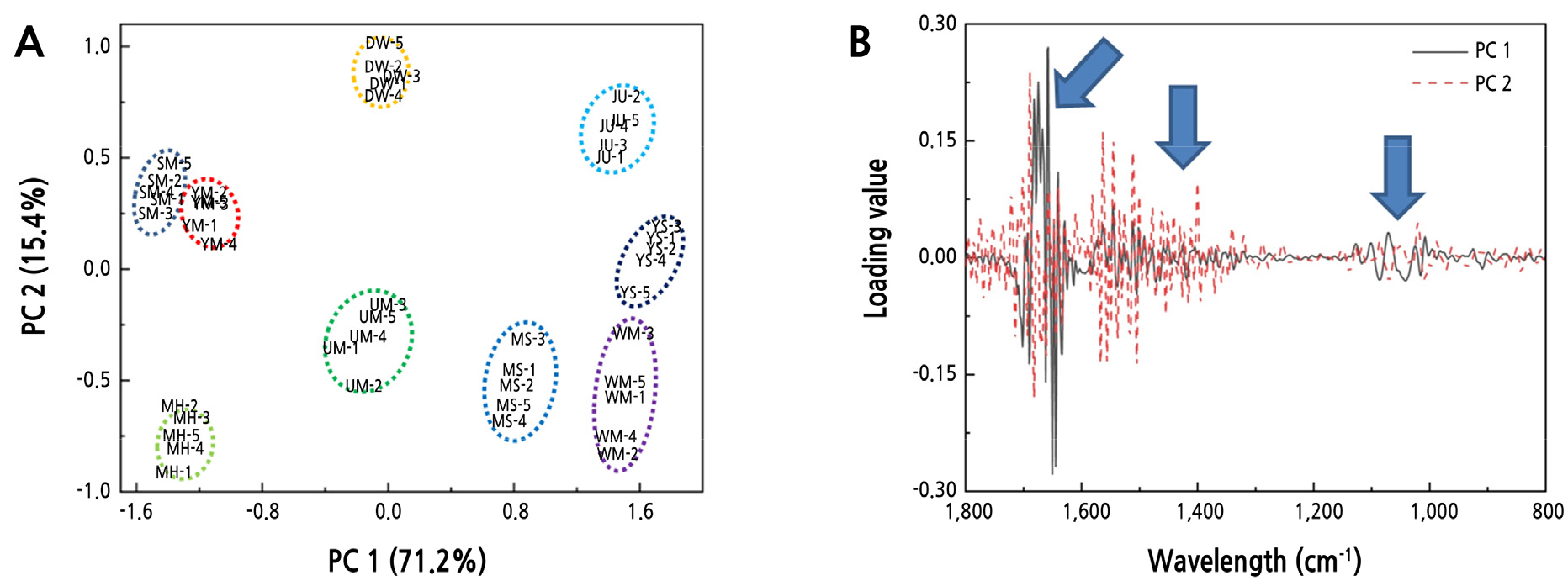
9품종 복숭아의 FT-IR 스펙트럼 데이터를 PCA 분석한 결과 PC 1과 2의 설명력이 71.2%와 15.4%로 각각 나타났다(Fig. 2A). ‘유명(UM)’을 중심으로 교잡 품종과 돌연변이 품종이 분포하는 것을 확인할 수 있었다(Fig. 2A). 또한, ‘치요마루(JU)’는 우측 상단에 위치하였고, ‘치요마루(JU)’ 품종이 교잡 품종과 돌연변이 품종에 비해 ‘유명(UM)’ 품종과의 유전적 유사성이 낮은 것으로 판단된다. ‘유명(UM)’의 자연 교잡 품종인 ‘음성(YS)’와 ‘대월(DW)’ 품종은 중앙 상단과 우측 중간에 위치하며 ‘유명(UM)’ 품종과의 유전적 유사성이 낮게 나타났고, ‘유명(UM)’의 돌연변이 품종인 ‘월미(WM)’ 품종은 우측 하단에 위치하면서 교배 품종인 ‘수미(SM)’에 비해 유전적 유사성이 낮게 나타났다. ‘유명 x 치요마루’ 교배종인 ‘유미(YM)’, ‘수미(SM)’, ‘미홍(MH)’ 그리고 ‘미스홍(MS)’ 품종은 ‘유명(UM)’ 품종의 좌, 우에 위치하였다. 이들 4품종 중 ‘유명(UM)’과 유전적 유사성이 가장 높은 것은 ‘미스홍(MS)’으로 나타났으며, ‘유미(YM)’와 ‘수미(SM)’ 품종이 유전적 유사성이 ‘미스홍(MS)’에 비해 낮게 나타났다.
복숭아 9품종의 genomic DNA 식별에 중요하게 작용한 FT-IR 스펙트럼 부위를 확인한 결과, PC 1과 2를 분류하는 중요한 기준은 1,700–1,500, 1,500–1,300 그리고 1,100–950cm-1 부위임을 알 수 있었다(Fig. 2B). 1,700–1,500, 1,500–1,300 그리고 1,100–950cm-1 부위는 FT-IR 스펙트럼상에서 차이를 나타냈던 부위(Fig. 1)와 일치하였으며, 이 부위가 나타내는 DNA 염기구조 C = O, C = N 그리고 C = C 형태와 –NH2결합 형태, guanine ring (C6 = C6), thymine ring (C4 = O4), uracil (C4 = O4), purine과 pyrimidine ring 구조, phosphodiester-deoxyribose backbone의 PO2- 그룹에 대한 대칭적 구조와 비대칭적 구조의 차이가 genomic DNA 식별에 중요한 역할을 하는 것으로 알 수 있었다(Schulz and Baranska, 2007)(Fig. 2B).
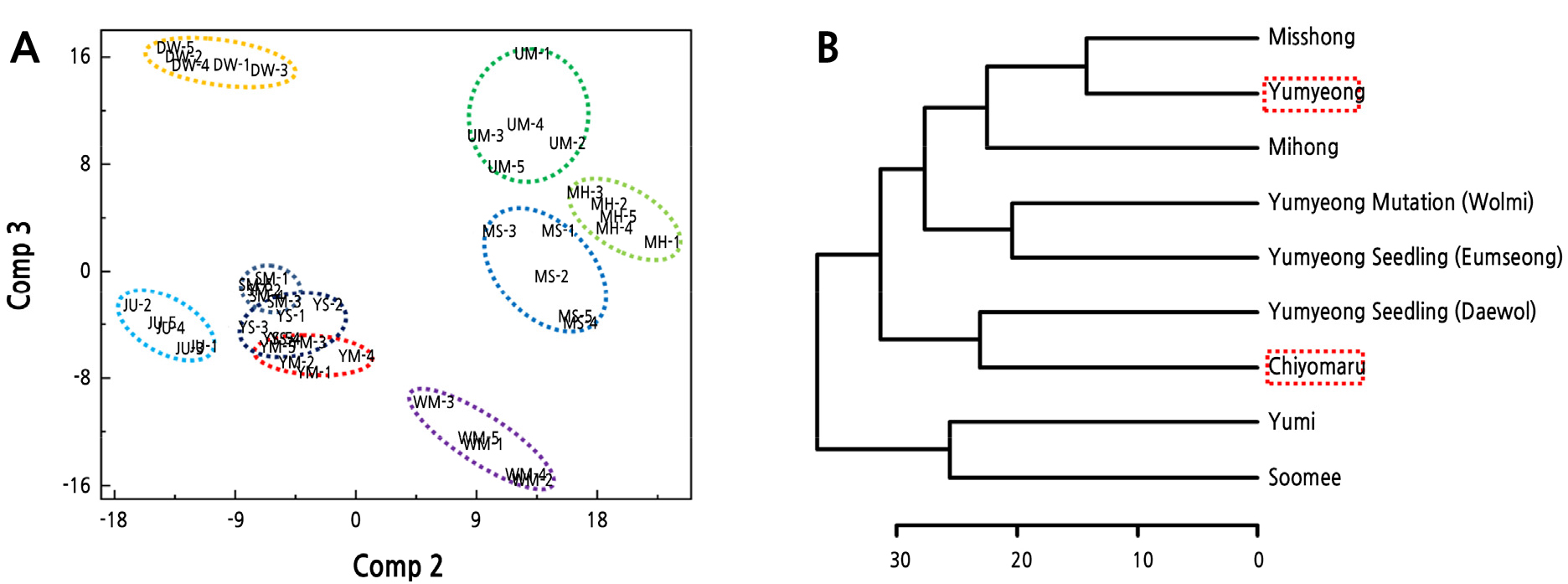
PLS-DA 분석의 경우 PCA 분석 보다 복숭아 품종간 차이가 뚜렷하게 나타났다(Fig. 3A). PLS-DA score plot을 보면 PCA와 동일하게 ‘치요마루(JU)’와 ‘유명(UM)’는 서로 좌우에 위치하면서 유전적 유사성이 낮은 것으로 나타났다. ‘치요마루(JU)’와 ‘유명(UM)’의 교배를 통해 얻어진 4품종 중 ‘미홍(MH)’, ‘미스홍(MS)’ 품종이 ‘유명(UM)’ 품종과 ‘유미(YM)’, ‘수미(SM)’ 품종이 ‘치요마루(JU)’과 유전적 유사성이 높은 것으로 각각 나타났으며 이 결과는 HCA dendrogram을 통해서도 확인할 수 있었다(Fig. 3B). 또한, ‘유명(UM)’의 돌연변이 품종인 ‘월미(WM)’ 품종은 교잡 품종에 비해 유전적 유사성이 낮은 것으로 나타났다(Fig. 3A). ‘유명(UM)’의 자연교잡실생인 ‘음성(YS)’은 ‘유미(YM)’, ‘수미(SM)’ 품종과 유연관계가 매우 높은 것으로 나타났으며, ‘대월(DW)’ 품종은 다른 품종에 비해 ‘유명(UM)’과의 유전적 유사성이 낮은 것으로 판단된다(Fig. 3A).
일반적으로 돌연변이 품종은 일부 염기서열에 변이가 일어나는 것이기 때문에 본래 품종과 유전적 정보가 매우 유사하며, Yamamoto(2003)에 따르면 복숭아 ‘아카츠키(Akatsuki)’와 그 돌연변이 품종 ‘효성(Gyousei)’을 17개의 SSR마커를 통해 분석하였을 때 다형성을 찾을 수 없었다. 한편 사과의 경우에는 분석법에 따라 아조변이를 구분하기도 하였는데 SSR로는 아조변이의 차이를 보이지 않았던 반면, Sequence-Specific Amplified Polymorphism(S-SAP) 분석으로는 그 차이를 확인할 수 있었다(Kuras et al., 2013). 하지만, FT-IR 스펙트럼을 이용한 분석은 SSR, S-SAP 등의 방법과는 다르게 특정 염기서열을 사용하여 분석하는 것이 아니라 파장별 흡광도를 비교하여 genomic DNA의 전체적인 구조를 분석하고 측정된 염기서열 중 달라지는 염기를 비교 분석하는 방법이다. 이번 실험에서 ‘유명’과 ‘월미’가 가장 큰 차이를 보인 파장대는 1,750–1,500cm-1 로 조사되었으며(Fig. 1), 이 파장대의 흡광도는 guanine (G), adenine (A), cytosine (C), uracil (U) 그리고 thymine (T)의 단일, 이중, 삼중 결합에 의해 결정된다. 특히 U와 T (C2 = O2 stretch of U, T)의 단일과 이중 결합이 나타나는 1,698–1,691cm-1 파장대에서 가장 극명한 차이를 보여 피리미딘 염기에 관련된 변화가 있을 것으로 추정되며 결합 값 등의 특정을 통해 복숭아 변이에 대한 분석이 가능할 것으로 예상된다(Banyay et al., 2003).
복숭아 품종간 Classification, prediction 그리고 cross-validation
각각의 복숭아로부터 품종간 PLS-DA 식별이 bootstrap test를 통해 실제 적용 가능성을 확인하였다(Table 2). Bootstrap test의 정확도는 99%로 통계적으로 유의하였으며(p < 0.01), RMSEP (Root Mean Square Error of Prediction)은 0.23, MSEP (Mean Square Error of Prediction)는 0.005로 예측값에 대한 정확도를 나타냈다(Table 2). 이러한 결과는 복숭아 품종간의 차이를 PLS 모델링을 통해 높은 정확도로 명확히 구별 할 수 있다는 것을 나타냈다. 본 연구에서는 9개의 복숭아 품종을 테스트하였고, 테스트를 통해 정확하게 예측하고 자신의 그룹으로 분류 되었다. Genomic DNA의 특정한 nucleoside base 그룹은 물리적인 진동으로 인해 변하기 쉬운 구조의 DNA이지만 서로 다른 FT-IR 스펙트럼의 특징적인 polypeptide-DNA 복합체 정보를 나타낼 수 있다고 보고되었다(Mello and Vidal, 2012). 따라서 품종간 예측 정확도를 통해 새로운 샘플이 사용되면 다양한 통계 연산을 사용하여 FT-IR 스펙트럼의 재현성을 나타낼 수 있다.
Table 2.
Summary of the PLS-DA classification results from FT-IR spectral data of genomic DNA. In bootstrapping, each case was classified by the functions derived from all other cases. DW : Yumyeong seedling (Daewol), JU : Chiyomaru, MH : Mihong, MS : Misshong, SM : Soomee, UM : Yumyeong, WM : Yumyeong mutation (Wolmi), YM : Yumi, YS : Yumyeong seedling (Eumseong)
| Prediction | Total | |||||||||||
| DW | JU | MH | MS | SM | UM | WM | YM | YS | ||||
| Cross validated | Count | DW | 5 | 5 | ||||||||
| JU | 5 | 5 | ||||||||||
| MH | 5 | 5 | ||||||||||
| MS | 5 | 5 | ||||||||||
| SM | 5 | 5 | ||||||||||
| UM | 5 | 5 | ||||||||||
| WM | 5 | 5 | ||||||||||
| YM | 5 | 5 | ||||||||||
| YS | 5 | 5 | ||||||||||
| Total | 5 | 5 | 5 | 5 | 5 | 5 | 5 | 5 | 5 | 50 | ||
복숭아 품종에는 매우 많은 종류가 있지만 유전적 다양성은 낮은 것으로 알려져 있다(Kwon et al., 2015). 복숭아는 Prunus persica라는 단일 종으로 구성 되어 있으며, 자가결실이 가능하다는 생물학적인 특징 이외에 육종 과정에서 반복적으로 유사한 품종을 모수로 활용되는 경우가 많기 때문에 유사한 유전자가 집적되고 있으며, SSR 마커 등 분자유전학적인 분석을 통해서도 유전적 다양성이 낮은 것으로 보고되고 있다(Xie et al., 2010). 현재까지 복숭아 genomic DNA의 FT-IR 스펙트럼 데이터를 이용한 복숭아에서 근연관계 분석에 대한 보고는 거의 없지만, 다른 작물에서는 genomic DNA의 FT-IR 스펙트럼 데이터 분석을 통해 품종 식별을 한 사례가 보고되어 있다. 특히, 적배추에서는 서로 다른 부모에서 F1 자손 식별이 가능하였고(Song et al., 2014), genomic DNA의 스펙트럼 유사성으로 고등 식물의 분류학적 관계를 나타냈으며(Muntean et al., 2009), FT-IR 스펙트럼의 유사성을 기준으로 차나무의 품종을 식별하였다(Song et al., 2017). 또한, 대목용 복숭아와 야생 복숭아의 유전적 유사도를 확인하고, 수집 지역에 따른 유전적 다양성이 낮은 것으로 보고된 바 있다(Cho et al., 2012). 이상의 연구 결과로 판단할 때 복숭아 genomic DNA의 FT-IR 스펙트럼 데이터를 이용한 근연관계 식별이 가능함을 알 수 있었다.
본 연구에서 확립된 genomic DNA를 이용한 복숭아의 품종 식별 기술은 품종, 계통의 신속한 선발 수단으로 활용이 가능할 것으로 기대되며 육종을 통한 품종개발 가속화에 기여 할 수 있을 것으로 예상된다.