서 언
토마토는 가지과 작물로 남아메리카 서부와 중부 에콰도르의 안데스 산맥이 원산지이다(Peralta and Spooner, 2006). 토마토에는 lycopene과 quercetin, phytoene, phytofluene, cyclolycopene, salicylates 그리고 tomatine과 같은 다양한 생리활성 물질들이 함유되어 있으며, 이들은 lycopene과 함께 항암작용에 관여하는 것으로 알려져 있다(Phyllis, 2004). 또한, 고부가가치 작물로 전세계적으로 생산량 및 재배면적이 꾸준히 증가하고 있는 채소작물이다. 2022년 FAOSTAT에 따르면 수출은 US 달러로 476억불, 수입은 2,946억불로 무역 가치가 지속적으로 증가하고 있어 경제적으로 중요한 작물이다.
기후변화와 더불어 바이러스, 박테리아, 그리고 균류와 같은 비생물학적 스트레스로 인해 토마토의 생산량이 감소되고 있다. 전세계적으로 식물병해충의 피해로 작물 생산량의 36%가 손실되며, 이 중 식물병원균에 의한 질병으로 전체 작물 수확량의 14% 정도가 감소된다. 또한, 다양한 질병 중 토양 매개 질병은 중요 식량 및 원예 작물 생산량에 대해 연간 10–20%의 수확량 손실이 발생한다(Nion and Toyota, 2015). 다양한 토양 전염성 병 중 하나인 풋마름병(Bacterial wilt)은 Ralstonia solanacearum(R. sol)에 의해 일어나며 식물의 뿌리에 침입하여 물관을 막아 시들음 증상을 유발하는 병이다(Genin, 2010). 이 병원균은 열대, 아열대 및 일부 온대 지역에 광범위하게 분포되어 있으며(Champoiseau et al., 2009) 기주 범위가 비정상적으로 넓고 토양 내에 장기간 생존할 수 있어 피해가 심각하다. 토양에서 월동하는 특성으로 인해 화학적 및 생물학적 방제가 어려우며(Lee et al., 2015b), 주로 윤작이나 저항성 품종 육성을 통한 저항성 유전자의 도입과 같은 병 발생을 억제하는 방향으로 연구가 진행되고 있다(Balatero et al., 2005; Lee et al., 2011; Kim et al., 2022).
바이러스 기반 유전자 침묵(Virus-Induced Gene Silencing, VIGS) 기술은 식물에서 다양한 유전자의 기능을 분석 할 수 있는 효과적인 방법이다(Ramegowda et al., 2014). 이 기술은 전사 후 유전자 침묵 현상(Post-Transcriptional Gene Silencing, PTGS)을 기반으로 내인성 유전자의 활성을 감소시키기 위해 재조합 바이러스를 사용하는 기술이다(Baulcombe, 1999). 바이러스에 감염된 식물은 바이러스 RNA를 분해하는 dsRNA(double strain RNA) 매개 PTGS를 유도한다. dsRNA는 DICER라는 효소에 의해 siRNA(small interfering RNA)로 절단되고 RISC 복합체(RNA-induced silencing complex)가 인식하여 세포 내에 상보적인 RNA 서열을 식별하고 분해를 통해 유전자의 발현이 억제된다(Waterhouse and Fusaro, 2006; Ding and Voinnet, 2007). 최근 유전체 분석을 기반으로 토마토, 고추를 비롯한 다양한 작물에서 후보 유전자 대량 규명(high-throughput screening)을 위해 분자표지 개발 및 VIGS 시스템이 효과적으로 적용되고 있다(Liu et al., 2002; Kim et al., 2019; Kang et al., 2021). 풋마름병 저항성 유전자 규명 연구에서도 유전학과 유전체를 기반으로 후보 유전자를 선발하고 VIGS를 통해 효과적으로 유전자 기능 규명에 활용할 수 있다(Kang et al., 2022). 하지만 VIGS를 활용한 유전자 침묵 기법의 경우 토마토, 담배에서는 각각 21°C 이하와 25°C 에서 가장 높은 효율을 보이며 이보다 높은 온도에서는 효율이 떨어진다(Burch-Smith et al., 2004). 반면, 풋마름병원균은 28–30°C에서 균의 증식과 병원성이 높은 것으로 보고 되었다(Lee et al., 2015a). 이로 인해 풋마름병원균과 유전자 침묵 기술의 최적화된 활성 온도들이 서로 상충 된다는 문제점이 있다.
R. sol은 온도 및 환경에 따라 병 발생 정도가 달라 R. sol과 VIGS 시스템 적용에 최적화된 온도 조건을 구축하여 풋마름병 저항성 후보 유전자의 기능 분석에 활용하고자 한다. 효율적인 활성온도 확인을 위해서 유전자 침묵을 유도하고 다양한 온도 조건을 확인하여 토마토의 생장과 유전자 침묵 효율 검증을 수행하였다. 이러한 연구 결과는 풋마름병 저항성 토마토의 유전자 침묵 효율 최적화를 통해 풋마름병 저항성 후보 유전자의 기능 분석에 효과적으로 활용될 것이며 다른 병원균의 저항성 유전자 탐색 실험에서의 적온 검정에 적용 될 것이다.
재료 및 방법
식물 재료 및 생육 환경
풋마름병 저항성 토마토 계통인 ‘Hawaii7996’을 사용하였으며 감수성 계통은 ‘Moneymaker’를 사용하여 실험을 수행하였다. 최아 후 암조건에 24시간 동안 28°C로 유지되는 인큐베이터에서 4–5일간 발아시켰고, 이후 광 16시간, 암 8시간 주기의 30°C로 유지되는 항온실에서 2–3일간 생육하였다. 발아 후 32공 트레이에 정식하여 이틀 간 광 16시간, 암 8시간 주기의 30°C 항온실에서 떡잎이 최대 생육에 이를 때까지 성장시켰다.
VIGS 접종 및 유전자 발현 확인
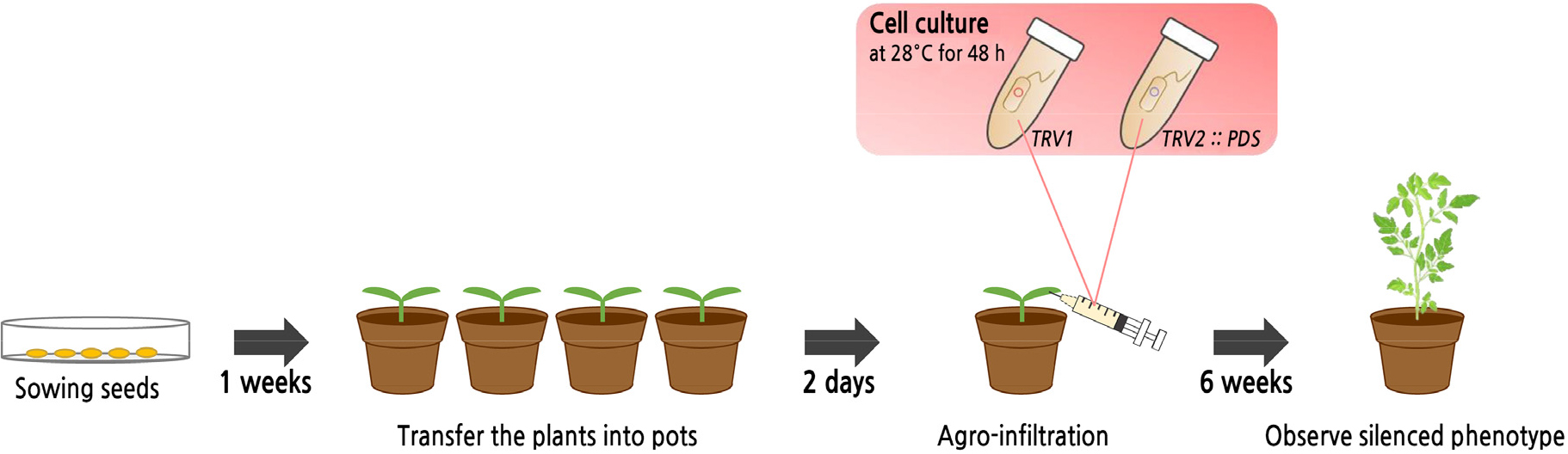
TRV-based VIGS 방법은 다음 두 가지의 논문(Yeom et al., 2012; Kang et al., 2018)의 실험과정을 참고하여 수행하였으며 pTRV1, pTRV2::PDS 벡터가 삽입된 플라스미드를 Agrobacterium tumefaciens sp. GV3101에 형질전환하여 사용하였다. 형질전환 Agrobacterium은 kanamycin 50mg·mL-1과 rifampicin 50mg·mL-1를 포함한 YEP(yeast extract peptone media)고체 배지에 streaking하여 28°C 암조건에서 48시간 동안 배양하였다. 자라난 균의 활성을 유지하기 위해 접종일까지 4°C 냉장고에 보관하였다. 배양된 Agrobacterium은 정식 2일 후 제1본엽이 나오기 전 떡잎이 최대 생장한 상태에서 접종을 수행하였다. 증식한 배양액을 원심분리하여 상층액을 제거하고 infiltration buffer(10mM MES, 10mM MgCl2, pH 5.5)로 침전된 세포를 용해시킨다. 세균현탁액에 infiltration buffer을 첨가하고 600nm에서 흡광도(optical density, OD)를 측정하여 0.5로 정량하였다. 접종액에 200µM의 acetosyringone을 분주하여 3–5시간 동안 실온에서 배양하여 접종액을 1mL 주사기를 사용하여 토마토의 떡잎에 접종하였다(Fig. 1).
Total RNA는 Trizol(Invitrogen, Carlsbad, USA)을 사용하여 추출하였으며 Superscript IV(Invitrogen, Carlsbad, USA)를 사용하여 2㎍의 total RNA를 complementary DNA(cDNA)로 합성하였다. PDS 유전자의 발현 정량화를 위해 quantitative RT-PCR을 수행하였다. 프라이머는 tPDS_F(5’-TGATGCAACGATGAAGGAAC-3’), tPDS_R(5’-CTGATAAGACAGCGCCTTCC-3’)을 사용하여 수행하였으며, 토마토 ubiquitin(UBI) 유전자의 발현을 대조구로 사용하였다(Mascia et al., 2010).
온도 환경 조절
접종 직후 Agrobacterium의 활성이 좋아지도록 16°C 암조건의 생장상(JSPC-420C, JS Research Inc., Gongju, Korea)에서 24시간 동안 저온 배양하였다. 이후 광 16시간, 암 8시간 주기의 생장상에서 5가지의 온도 처리로 나누어 4주 동안 실험을 진행하였다. 접종 후 4주에 유전자의 침묵 효율을 조사했으며 SAS 프로그램(SAS 9.4, SAS Institute Inc., Cary, NC, USA)을 사용하여 통계분석을 수행하였다. 분산분석(ANOVA)을 수행하였으며 Tukey의 다중비교법을 통해 분산분석 간의 평균을 비교했다.
풋마름병원균 접종 및 발병 조사
R. solanacearum SL341 균주는 동아대학교에서 분양 받아 실험에 사용하였다(Jung et al., 2014). 풋마름병원균주를 2,3,5-triphenyl tetrazolium chloride(TZC)가 포함된 CPG 고체 배지에 도말하여 28°C에서 48시간 배양하였다. 이후 배양된 병원균을 CPG 액체 배지에 넣어 250rpm으로 28°C에서 24시간 배양하였다. 세균 배양액을 상온에서 10,000rpm 5분간 원심분리 후, 상층액을 제거하고 회수한 세균 pellet에 멸균수를 넣어 희석 후 현탁액을 만들었다. VIGS 접종 후 28일이 되는 시점의 식물체에 R. solanacearum SL341을 107 CFU/soil(OD600 = 0.3) 농도로 관주(root drench) 접종하였다. 접종 개체는 모두 30°C 생장상에서 광 16시간, 암 8시간 주기로 20일간 병 평가를 수행하였다. 풋마름병 발병 조사는 토마토 전체 잎에서 시든 정도에 따라 0 = 풋마름병 증상 없음, 1 = 전체 잎의 1–25% 시들음, 2 = 전체 잎의 26–50% 시들음, 3 = 전체 잎의 51–75% 시들음, 4 = 76–100% 시들음 총 5단계의 점수로 병의 진전을 확인하였다(Jung et al., 2014). 모든 실험은 각 처리구 별로 7개체씩 총 3반복을 수행하였으며, SAS 프로그램(SAS 9.4, SAS Institute Inc, Cary, NC, USA)을 이용하여 분산분석(ANOVA)을 수행하고 Tukey의 다중비교법을 통해 분산분석 간의 평균을 비교했다.
결과 및 고찰
Agrobacterium을 매개로 VIGS를 수행한 후 16°C 암조건의 생장상에서 하루 동안 저온 배양하였으며 풋마름병 저항성 토마토의 유전자 침묵 효율 환경 최적화를 위해 다음과 같은 방법으로 생장상에서 4주 동안 실험을 진행하였다. 저온은 22°C, 고온은 30°C를 처리하였으며 1) 4주간 저온(4L), 2) 1주 저온 후 3주 고온(1L3H), 3) 2주 저온 후 2주 고온(2L2H), 4) 저온 3주 후 고온 1주(3L1H), 5) 4주간 고온(4H)으로 총 다섯 가지의 처리로 온도를 조절하여 수행하였다(Fig. 2). 풋마름병과 유전자 침묵 기법에서의 가장 효율적인 온도를 선발하는 기준은 Agrobacterium 매개의 VIGS 적용과 풋마름병원균을 접종하기에 식물체가 충분히 크며 균 활성에 적절한 온도이여야 한다. 따라서 VIGS 접종 후 풋마름병원균을 접종하기에 적합한 식물체 크기와 유전자 침묵 효율에 적합한 온도 환경을 확립하기 위해 위 조건에 따른 R. solanacearum 접종 스크리닝을 수행하였다.
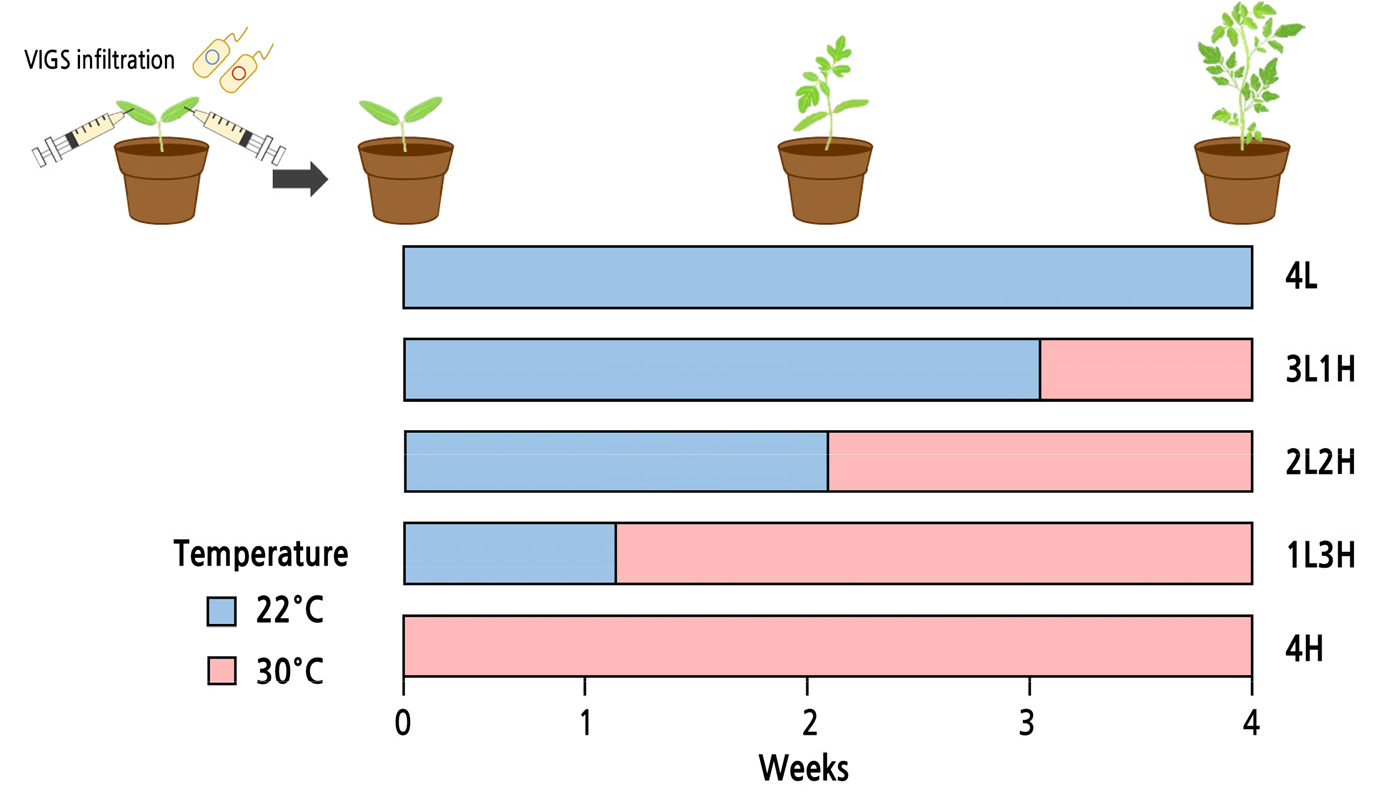
Fig. 2.
Five temperature conditions for optimization of the silencing efficiency in a Ralstonia solanacearum resistant tomato cultivar. Schematic diagram of five temperature conditions for four weeks after VIGS infiltration. Light blue and light pink bars indicate the low temperature (22°C, L) and high temperature (30°C, H) conditions. 4L: low temperature for four weeks, 3L1H: three weeks at a low temperature and one week at a high temperature, 2L2H: two weeks at a low temperature and two weeks at a high temperature, 1L3H: one week at a low temperature and three weeks at a high temperature, 4H: high temperature for four weeks.
풋마름병 접종 실험을 하기 위해서는 식물체가 3–4엽이 최대 신장을 이루며 7–8엽이 출현하고 있어야 하며 접종 후 28–30°C를 유지해주어야 풋마름병원균이 안정적으로 활성을 나타낸다(Kunwar et al., 2020; Kwon et al., 2021). 반면 바이러스 기반 유전자 침묵 기법에서는 agro-infiltration 이후 암조건에서 16°C로 유지되는 챔버에서 24시간 동안 배양 후 20–22°C에서 생장 시 안정적인 유전자 침묵 효율을 나타낸다. 우선 풋마름병을 접종하기에 적합한 식물체의 크기를 나타내는 온도 환경은 3L1H, 2L2H, 그리고 1L3H 식물체임을 확인 할 수 있었다(Fig. 3). 4L은 풋마름병을 접종하기에 3–4엽이 최대를 이루지 않았으며 4H는 9–10엽이 최대 생장을 이룰 정도로 많이 성장하여 풋마름병에 대한 정확한 표현형을 나타낼 수 없다고 판단하였다.

Fig. 3.
Confirmation of a PDS-silencing phenotype suitable for R. sol inoculation under five different temperature conditions. A. Phenotype of PDS-silenced Hawaii7996 at various temperatures. PDS-silenced tomato plants under two different temperatures (22°C and 30°C). B. Measurement of the height of PDS-silenced plants in five different temperature growth conditions. L and H indicate the low temperature (22°C) and high temperature (30°C) conditions, respectively. The experiment in Fig. 3 was conducted with four biological replicates, with each containing four tomato seedlings. Vertical bars indicate the standard errors of the mean (n = 4). The results of Tukey's multiple range test are also shown (p < 0.05, n = 4).
다음으로 유전자 침묵 효율에 적합한 온도 환경을 확립하기 위해서 접종 4주 후, 식물체의 3–6번 본엽에서 다음과 같은 기준으로 조사하였다. 유전자의 침묵 정도에 따라 0 = 침묵 되지 않음, 1 = 면적이 1–25% 침묵, 2 = 26–50% 침묵, 3 = 51–75% 침묵, 4 = 76–100% 침묵 된 것으로 총 5단계의 기준으로 표현형을 확인하였다(Fig. 4A). 이때 3점 이상인 식물체는 유전자 침묵이 되었다고 판단하였으며 0–2점은 침묵이 되지 않은 것으로 간주하였다. 유전자 침묵이 충분히 유도되었다고 판단되는 처리구는 4L, 3L1H, 그리고 2L2H 식물체임을 확인했다(Fig. 4B). 4H 식물체는 전체적으로 유전자 침묵이 불균일하게 발생하였으며, 1L3H는 4H를 제외한 처리구 중 가장 낮은 유전자 침묵 효율을 나타내었다. 추가적으로 유전자 발현 확인을 통해 유전자의 침묵 정도를 수치화하여 나타내었다(Fig. 4C). 모든 처리구에서 PDS(phytoene desaturase) 유전자가 침묵 되었으나 대조구인 TRV2::GFP와 비교하였을 때 4L, 3L1H, 그리고 2L2H에서 PDS 유전자의 침묵 효율이 높았으며 이와 대조적으로 1L3H와 4H는 PDS 유전자의 침묵 효율이 낮았다. 1L3H와 4H는 장시간의 고온 처리로 인해 낮은 침묵 효율이 나타났으며 풋마름병 저항성 후보유전자 발굴을 위해 활용하기에는 적합하지 않다고 판단하여 적정 온도 대상에서 제외하였다. 또한, 풋마름병원균 접종 8일차에 1L3H 처리구는 저항성 대조구인 ‘Hawaii7996’과 비교하였을 때 생존율이 낮아 풋마름병원균의 반응성 확인에 적합하지 않다고 판단하였으며 4L과 4H 처리구는 풋마름병균 접종에 적합하지 않은 크기로 제외하였다(Suppl. Fig. 1).
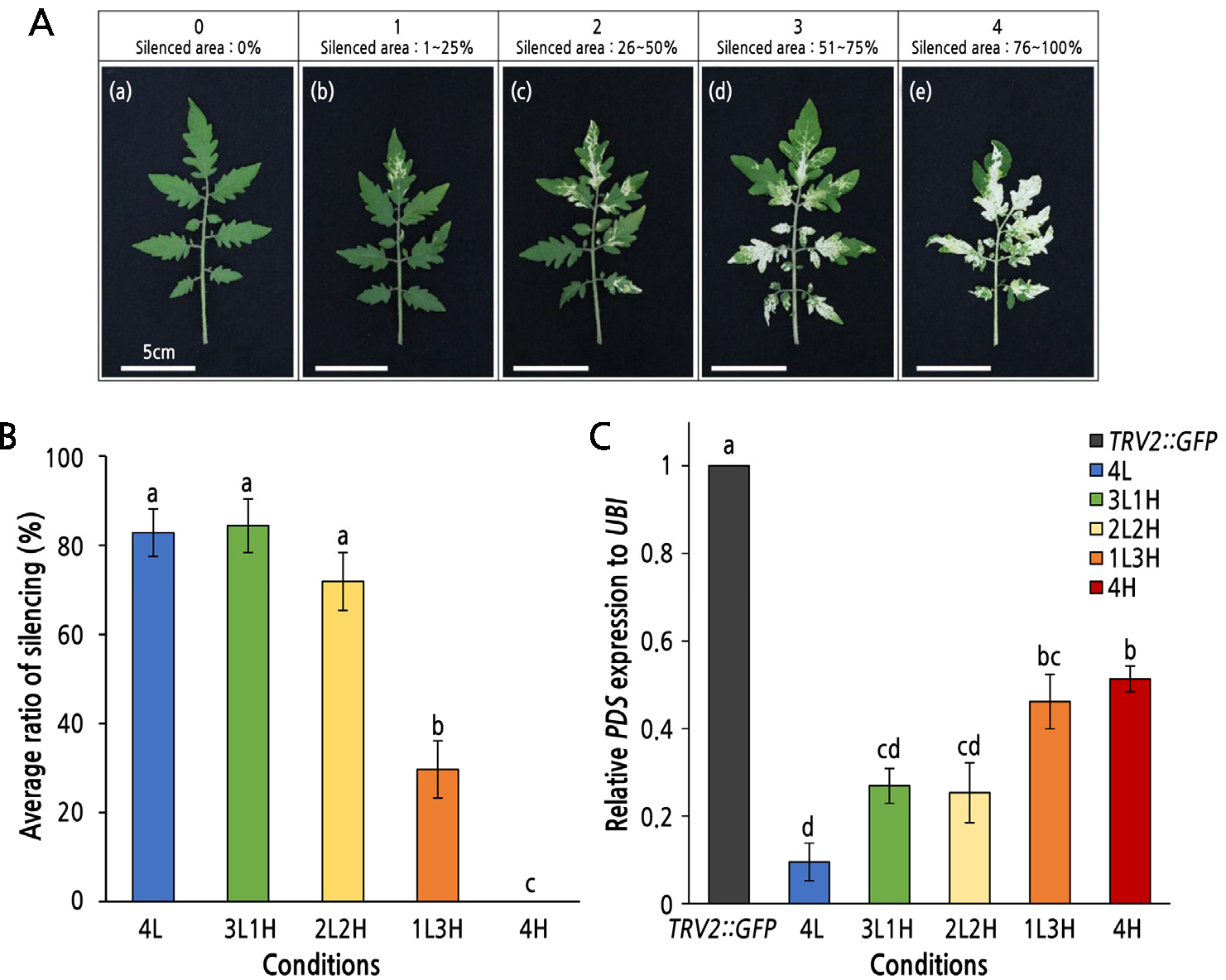
Fig. 4.
PDS-silencing efficiency of leaves under various temperatures. A. Scoring criteria of the silencing efficiency. Silencing was evaluated using five scoring categories: (a) 0, no silencing; (b) 1, slight silencing (1-25%); (c) 2, moderate silencing (26–50%); (d) 3, substantial silencing (51-75%); and (e) 4, severe silencing (76–100%). The silencing efficiency was calculated based on the surface area using the five categories. B. The graph shows the average ratio of PDS silencing under the five different temperature conditions. The areas of photo-bleached leaves as measured in the third to sixth true leaves in four tomato plants are shown. The experiments in Fig. 4A and 4B were conducted with four biological replicates, and each contained four tomato seedlings. Vertical bars represent the standard errors of the mean (n = 4). The results of Tukey's multiple range test are also shown (p < 0.05, n = 4). C. Relative expression of PDS to ubiquitin in different temperature conditions. Vertical bars indicate the standard errors of the mean (n = 3). Different letters in the column indicate significant differences as determined by Tukey's multiple range test (p < 0.05, n = 3). TRV2::GFP: control, 4L: the low temperature for four weeks, 3L1H: three weeks at a low temperature and one week at a high temperature, 2L2H: two weeks at a low temperature and two weeks at a high temperature, 1L3H: one week at a low temperature and three weeks at a high temperature, 4H: high temperature for four weeks.
풋마름병 저항성 후보 유전자 발굴에 활용 가능한 토마토의 적정 접종 환경을 확립하기 위해 풋마름병원균을 관주하여 유전자 침묵 개체의 풋마름병 발병을 평가하였다(Fig. 5). 감수성 품종인 ‘Moneymaker’는 접종 후 5일에서 6일 사이에 발병을 시작하여 8일차에 고사하는 전형적인 감수성 양상을 나타냈다(Abebe et al., 2020). 반면, 2주 저온 후 2주 고온 생육한 2L2H 식물체는 ‘Hawaii7996’과 3주 저온 후 1주 동안 고온 생육한 3L1H 개체와 비교하였을 때 12일 차부터 발병을 시작하여 13일차부터 점차적으로 고사하였다. 따라서 결과를 종합해보면 저항성 대조구인 ‘Hawaii7996’과 유사한 양상을 보이는 3L1H 식물체가 유전자 침묵 기법과 풋마름병 접종을 함께 적용하기에 가장 적합한 생육조건이라 판단하였다. 풋마름병원균에 대한 저항성은 양적형질 유전자좌에 의해 조절되며 온도 및 생육 조건과 같은 환경에 의해 저항성 반응이 달라져 저항성 후보 유전자 발굴에 어려움이 있다(Kunwar et al., 2020). 본 연구를 통해 온도 처리 기간에 따른 적합한 생육조건을 찾음으로써 추후 토마토의 풋마름병 저항성 관련 유전자를 분석하고 이에 대한 기능 분석 연구의 기반 자료로 활용 가능할 것이다.
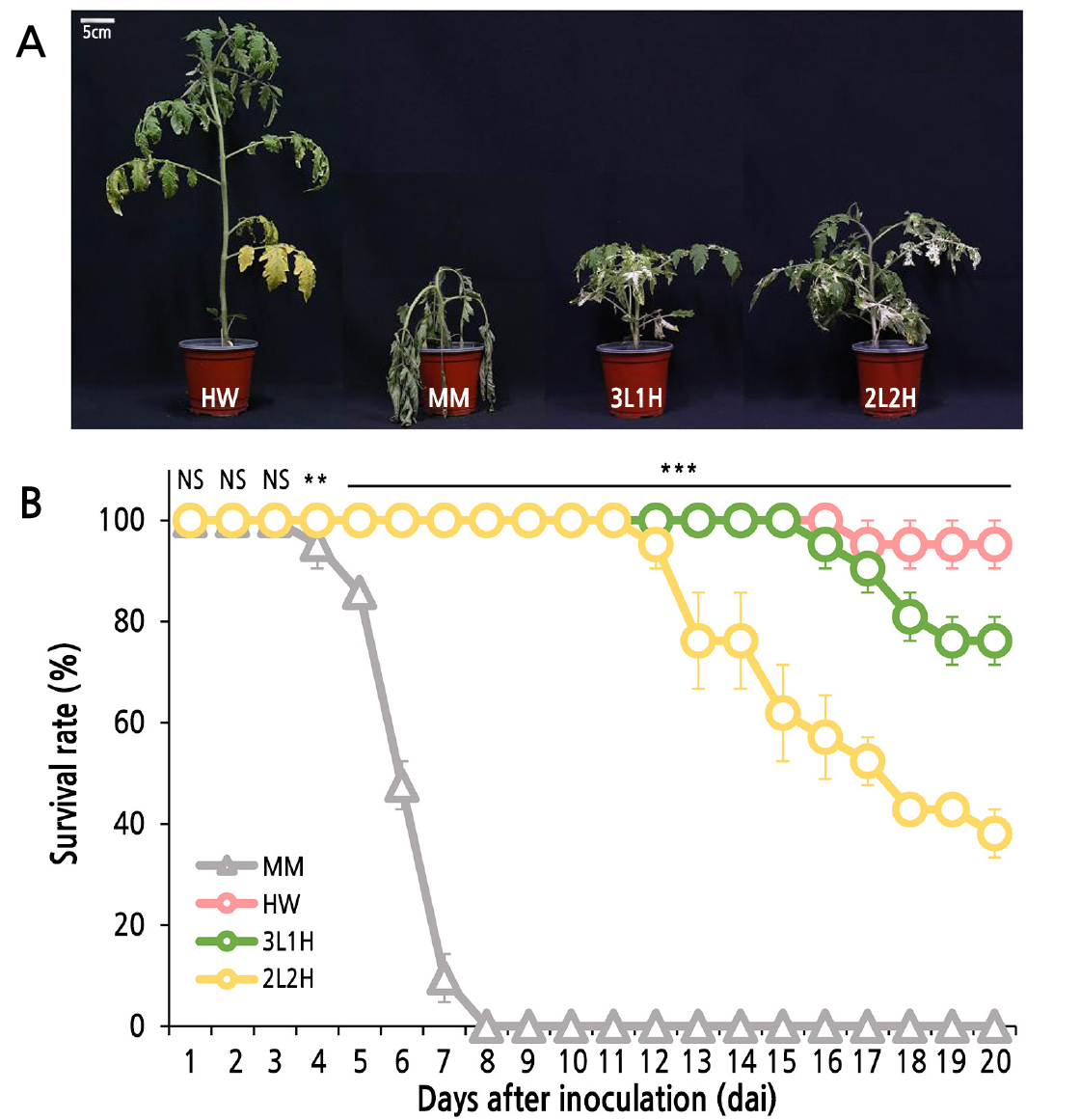
Fig. 5.
Evaluation of bacterial wilt symptoms against R. solanacearum in VIGS tomatoes. In this study, 28 days after VIGS, the tomatoes were inoculated with R. solanacearum SL341 by root drenching with a bacterial suspension of 1 × 107CFU/soil (OD600 = 0.3). The plants were grown in a chamber at 30°C with a 16h light and 8h dark cycle. MM: R. sol susceptible cultivar ‘Moneymaker’, HW: R. sol resistant cultivar ‘Hawaii7996’, 3L1H: three weeks at a low temperature and one week at a high temperature, 2L2H: two weeks at a low temperature, and two weeks at a high temperature. A. Wilting symptoms against R. solanacearum SL341 on control plants and PDS-silencing plants at six days after inoculation. B. The progress degree of the survival rate against R. solanacearum on control tomatoes and PDS-silenced ‘HW7996’ (3L1H and 2L2H). Vertical bars represent the standard errors of the mean (n = 3). The results of Tukey's multiple range test are also shown (p < 0.05, n = 3). NS, **, and *** indicate non-significant or significant at p < 0.01 or 0.001, respectively.
Supplementary Material
Supplementary materials are available at Horticultural Science and Technology website (https://www.hst-j.org).
- HORT_20230039_Figure_1s.pdf
Survival rate of PDS-silenced tomato against R. solanacearum. This graph shows the survival rate of R. sol inoculated tomato plants at eight days after inoculation. MM: ‘Moneymaker’ as a susceptible control, HW: ‘Hawaii7996’ as a resistant control, 4L: low temperature for four weeks, 3L1H: three weeks at a low temperature and one week at a high temperature, 2L2H: two weeks at a low temperature and two weeks at a high temperature, 1L3H: one week at a low temperature and three weeks at a high temperature, and 4H: high temperature for four weeks.