서 언
재료 및 방법
식물재료
과피 색도 측정
유리당 함량 분석
RNA 추출, cDNA 라이브러리 구축 및 염기서열 분석
전사체 de novo Assembly
전사체 Functional Annotation
DEGs 분석
통계처리
결과 및 고찰
단감 과실 발육 단계별 색도 변화
단감 과실 발육 단계별 유리당 함량 변화
전사체 de novo Assembly
전사체 Functional Annotation
DEGs 분석 및 Clustering
탄수화물 대사 관련 유전자 발현 양상
카로티노이드 생합성 관련 유전자 발현 양상
성숙 조절 전사인자 발현 양상
서 언
국내 단감 재배면적의 품종별 비율은 ‘부유’ 82.7%, ‘차랑’ 9.2%, ‘서촌조생’ 3.2%, 기타 4.9%로(KREI, 2018), 만생종인 ‘부유’ 품종의 편중재배가 이루어지고 있으며, 이로 인해 수확기 서리피해와 동해, 노동력의 일시 집중, 홍수 출하에 의한 가격 하락 등의 문제가 발생하고 있다. 따라서, 단감의 국내 소비시장에서는 수확 및 유통시기를 분산할 수 있는 조생종 또는 중생종 품종이 요구되고 있으며, 아울러 추석 전에 수확하여 선물용이나 제수용으로 사용 가능하면서 소비자가 선호하는 고품질의 새로운 완전단감 품종이 필요한 상황이다.
과실의 생장 및 발육에 관련된 유전자로써 fw2.2는 토마토 과실 크기의 변화에 핵심적인 요소이며, fw2.2 gene family member인 cell number regulator(CNR)는 복숭아, 체리, 매실, 살구 등의 벚나무속 재배종 과실의 세포수와 기관 크기를 조절하는데 결정적인 역할을 한다(Frary et al., 2000; De Franceschi et al., 2013). 또한 mitogen-activated protein kinase kinase kinase(MAPKKK) gene family는 애기장대의 세포 분열과 생장을 조절한다(Krysan et al., 2002). 과실의 성숙기를 조절하는 것으로 알려진 전사인자(Transcription Factor)로는 N-terminal NAC domain과 C-terminal variable transcription regulatory regions(TRRs)로 이루어진 NAM, ATAF1/2, CUC2(NAC) 등이 보고되었는데(Zhu et al., 2015), 바나나의 경우 MaNAC1과 MaNAC2가 ethylene signaling pathway의 ethylene insensitive like(EIL)와 상호작용하여 과실 성숙을 조절한다(Shan et al., 2012). 또한, Pirona et al.(2013)은 NAC가 복숭아의 성숙기를 조절하는 qMD4.1 locus의 후보유전자임을 제시하였고, 오일팜, 토마토 등에서도 NAC가 과실 성숙에 관련되어 있음이 보고된 바 있다(Kou et al., 2016; Tranbarger et al., 2017).
한편, 전사체분석을 통한 연구에서는 Janssen et al.(2008)은 사과의 꽃눈에서부터 과실의 성숙시기까지 핵심 세포주기 유전자, 전분대사 관련 유전자, 과실 발육 후보유전자의 발현에 대해 보고하였고, 베이베리, 포도, 배, 매실 등에서도 과실의 발육 및 성숙에 대한 전사체 연구가 이루어진 바 있다(Feng et al., 2012; Sweetman et al., 2012; Xie et al., 2013; Kim et al., 2015). 감에서는 Habu et al.(2016)의 일본감 ‘평핵무’와 이의 아조변이체인 소과종 ‘Totsutanenashi’의 unigenes 발현수준을 이용한 gene set enrichment 분석 결과, 세포주기와 관련된 유전자 중에서 MAPKKK와 cyclin-D3-2가 두 품종 간에 차등적으로 발현되었다. 아울러 당 대사 및 탄닌 생합성 경로 유전자(Matsui and Kitagawa, 1988; Senter et al., 1991; Nakagawa et al., 2008; Akagi et al., 2009), 카로티노이드 생합성 경로 유전자(Niikawa et al., 2007; Nakagawa et al., 2008; Akagi et al., 2009)에 대한 연구가 이루어졌다. 하지만 감 특히, 단감의 과실 성숙을 통제하는 핵심 유전자 및 전사인자에 대해 고찰한 연구는 없었다.
감에서는 RNA sequencing(RNA-seq)을 이용한 전사체 분석(Kim et al., 2017)을 통해 aldehyde dehydrogenase(ALDH) 2 유전자가 alcohol dehydrogenase(ADH), pyruvate decarboxylase(PDC) 유전자와 함께 감 탈삽(Yoo et al., 2019; Win et al., 2019)에 중요한 기능을 한다는 것과, miR395p-3p와 miR858b가 각각 basic helix-loop-helix(bHLH)와 myeloblastosis (MYB)를 조절함으로써 감의 탈삽을 조절하는 miRNAs임이 보고되었으며(Luo et al., 2014; Luo et al., 2015), Jung et al.(2017)이 ‘부유’의 신선과와 연화과 과피의 RNA-seq을 통해 차등적으로 발현되는 유전자를 조사한 결과, 과실의 연화가 진행됨에 따라 당 대사에 관련된 유전자 DkUGD1, DkChiA3, DkB-Chi1과, 에틸렌 생합성 유전자인 1-aminocyclopropane- 1-carboxylate(ACC) synthase(DkACS1, -2, -3)와 ACC oxidase(DkACO1, -2)는 상향 조절되고, DkCAB1, DkFNR1, DkPsbY 등 광합성 경로 유전자는 하향 조절됨이 밝혀졌다. 그러나 현재까지 감에서 과실발육 및 성숙에 따른 유전자 발현량의 변화에 대한 포괄적인 전사체 분석은 이루어지지 않았다.
따라서 본 연구는 완전단감 품종 중에서 만생종인 ‘부유’와 조생종인 ‘조추’ 과실 성숙의 주요 지표가 되는 형질과, 발육 단계 및 품종 간 차등적으로 발현되는 유전자에 대한 전사체를 비교 분석하고 만생종과 조생종 간 숙기 차이에 결정적 요소가 되는 유전자를 탐지하고, 숙기와 밀접하게 연관된 분자마커 개발에 필요한 기반 자료 구축을 위해 실시하였다.
재료 및 방법
식물재료
만생종 완전단감 ‘부유’(Diospyros kaki Thunb. cv. Fuyu)와 조생종 완전단감 ‘조추’(Diospyros kaki Thunb. cv. Soshu)를 실험재료로 사용하였다. 경상남도농업기술원 단감연구소(경남 김해시 진영읍)의 사양토 평지 과원(북위 35° 16' 56", 동경 128° 42' 59")에 3 × 2m 거리로 재식된 10년생 ‘부유’(Fuyu)와 ‘조추’(Soshu)를 대상으로 품종별로 크기가 비슷하고 수세가 안정된 나무를 반복당 3주씩 완전임의 3반복으로 시험수를 배치하였다.
품종별 3나무를 임의로 선택하여 개화 후 9주부터 18주까지 3주 간격으로 나무당 3개의 과실을 채취하였다. 과실 표면을 70% 에탄올로 소독 후 꽃받침 부위를 제거하고 과실을 4등분하였다. 과실당 1/4 조각에서 과심과 종자를 제거하고 과육(과피 포함)을 2mm 정도의 크기로 주사위 모양으로 자른 후 1g을 취하여 나무별로 시료를 풀링하였다. 액체 질소에 시료를 즉시 얼린 후 RNA를 추출할 때까지 -70°C에 보관하였다.
과피 색도 측정
과피의 색도는 과실 적도 부위에서 평균적인 색깔을 띠는 부위를 선정하여 휴대용 색차계(CM-2500d, Konica Minolta Sensing Inc., Sakai, Japan)를 사용해서 과실당 1회씩 측정한 후 Hunter a/b 값을 구하였다.
유리당 함량 분석
유리당 함량 분석은 분쇄한 과육 10g에 3차 증류수 40mL을 가하여 30분간 추출한 뒤, 13000rpm으로 5분간 원심분리한 상층액을 0.45µm membrane filter로 필터링 후, 1mL을 취하여 carbohydrate column(7.8 × 300mm Cartridge, Waters, Milford, USA)을 장착한 HPLC(2690 Separations Module, Waters, Milford, USA)를 사용하여, 45°C로 설정한 column에 20µL씩 주입하여 분석하였다. 이동상은 acetonitrile과 water(80:20, v/v)로 유량은 분당 1.0mL였으며, 검출은 refractive index(RI) detector를 사용하였다. 시료와 같은 조건에서 총 3종의 유리당(fructose, glucose, sucrose) 표준물질을 분석하여 머무름 시간을 비교해 확인하였고, 각각의 검량곡선을 통해 함량을 산출하였다.
RNA 추출, cDNA 라이브러리 구축 및 염기서열 분석
총 RNA는 Ribospin Seed/Fruit RNA mini kit(317-150, GeneAll Biotechnology Co., Seoul, Korea)를 사용하여 추출하였다. RNA 샘플의 품질은 1.0% 아가로스젤 전기영동으로 평가하였고, RNA의 농도와 순도를 NanoDrop spectrophotometer (ND-1000, NanoDrop Technologies Inc., Wilmington, USA)를 사용하여 측정하였다. RNA 샘플의 integrity는 Agilent 2100 Bioanalyzer를 사용하여 측정하였으며. RNA integrity number(RIN) 값이 7 이상이고, 28s/18s 값이 1-3인 RNA 샘플을 후속 분석에 사용하였다.
RNA-Seq paired end library는 Illumina TruSeq RNA Sample Preparation Kit v2(RS-122-2001, Illumina Inc., San Diego, USA)를 사용하여 제작하였다. Poly(A) selection을 통해 총 RNA를 mRNA로 정제한 다음, RNA를 fragmentation buffer를 사용해서 단편화하고, random hexamer primer를 사용하여 first-strand cDNA 단편으로 전사시켰다. 그 다음, second strand cDNA를 만들어서 이중가닥 cDNA를 제작하였다. Library 제작은 ds-cDNA로부터 blunt-end cDNA 단편의 생성으로 시작하였다. 이후 blunt-end에 Adenine nucleotide(A)-base를 추가하여 단편을 시퀀싱 어댑터에 연결할 수 있도록 하였다. ligates의 size selection 후, 어댑터 시퀀스를 포함하는 ligated cDNA 단편을 adapter specific primer를 사용한 PCR을 통해 증폭하였다.
이 후 KAPA library quantification kit(KK4854, Kapa Biosystems Inc., Wilmington, USA)를 사용하여 library를 정량하고, 100bp paired-end sequencing reads로 만든 각 library를 Illumina Hiseq 2000 장비를 사용하여 high-throughput sequencing하였다.
전사체 de novo Assembly
100bp paired-end reads로 구성된 raw sequencing data를 phred quality score(Q) > 20와 read length > 25bp를 기준으로 필터링하였다. k-mer size와 어셈블리된 contig를 평가하기 위해 Velvet(v1.2.07) 소프트웨어를 사용하여 de novo assembly를 최적화하였다. Contig는 Oases(v0.2.08)를 사용하여 transcript isoform으로 병합하였다. Velvet과 Oases 프로그램 분석은 de Bruijn graph algorithm을 기반으로 하였다.
최적의 de novo assembly를 선택하기 위해 8가지의 hash length(k-mer = 51, 55, 57, 59, 61, 63, 65, 71)를 사용하여 초기 assembly를 수행하고, k-mer의 변화에 따른 transcripts의 total length의 변화가 적고, max length, average length, N50을 고려하여 편차가 적은 k-mer를 최적의 k-mer로 선정하였다.
전사체 Functional Annotation
각 샘플에서 취한 clean reads를 사용하여 단감 유전자의 어셈블리된 transcripts를 기능적으로 파악하기 위해, NCBI NR의 viridiplantae database(DB), phytozome DB(version 9.1)(http://www.phytozome.net/), Uniprotkb, EuKaryotic Orthologous Groups(KOG), 그리고 Kyoto Encyclopedia of Genes and Genomes(KEGG)의 amino acid sequence 정보에 BLASTX (filter 기준: e-value < 1e-10, Best hits)하여 annotation을 수행하였다. InterProscan은 EMBL에서 제공된 tools를 이용하였으며 default로 annotation을 하였다. Gene Ontology(GO) DB에서 제공하는 서열을 이용하여 alignment를 수행하였고, thresholds는 기능별 유전자의 총 수가 counts > 1 이상을 사용하였으며, GO depth는 1로 지정하여 3가지 functional category인 Biological Process(BP), Cellular Component(CC), Molecular Function(MF)로 분류하였다.
DEGs 분석
동일 품종별 과실 발육 단계간, 그리고 동일 과실 발육 단계별 ‘부유’와 ‘조추’ 간 차등적으로 발현된 유전자(Differentially Expressed Genes: DEGs)를 확인하기 위해, 두 개의 분석 세트를 구성한 후 bowtie2(v2.1.0) 프로그램(Langmead et al., 2009)을 사용해서 분석하였다. 세트 Ⅰ은 개화 후 9주차(9 week after bloom, WAB) 의 각 품종 샘플을 대조구로 하고, 이후 3주 간격(12, 15, 18WAB)의 샘플들을 처리구로 해서 총 6개 비교조합을 분석하였고, 세트 Ⅱ는 각 단계별로(9, 12, 15, 18WAB) ‘부유’를 대조구로, ‘조추’를 처리구로 해서 총 4개 비교조합을 분석하였다. Fold change 값이 2 이상인 것을 차등적 발현의 기준으로 하여 두 품종의 library에 대한 시퀀싱 결과를 짝비교하였고, p-value < 0.001, false discovery rate(FDR) < 0.01, log2 fold change > 2인 것을 유의한 값으로 지정하였다.
통계처리
단계별로 각 품종의 3반복으로부터 수집한 표현형 조사 성적을 EXCEL 프로그램을 활용하여 평균과 표준오차(SE)로 나타내어 두 품종 간 비교하였다.
결과 및 고찰
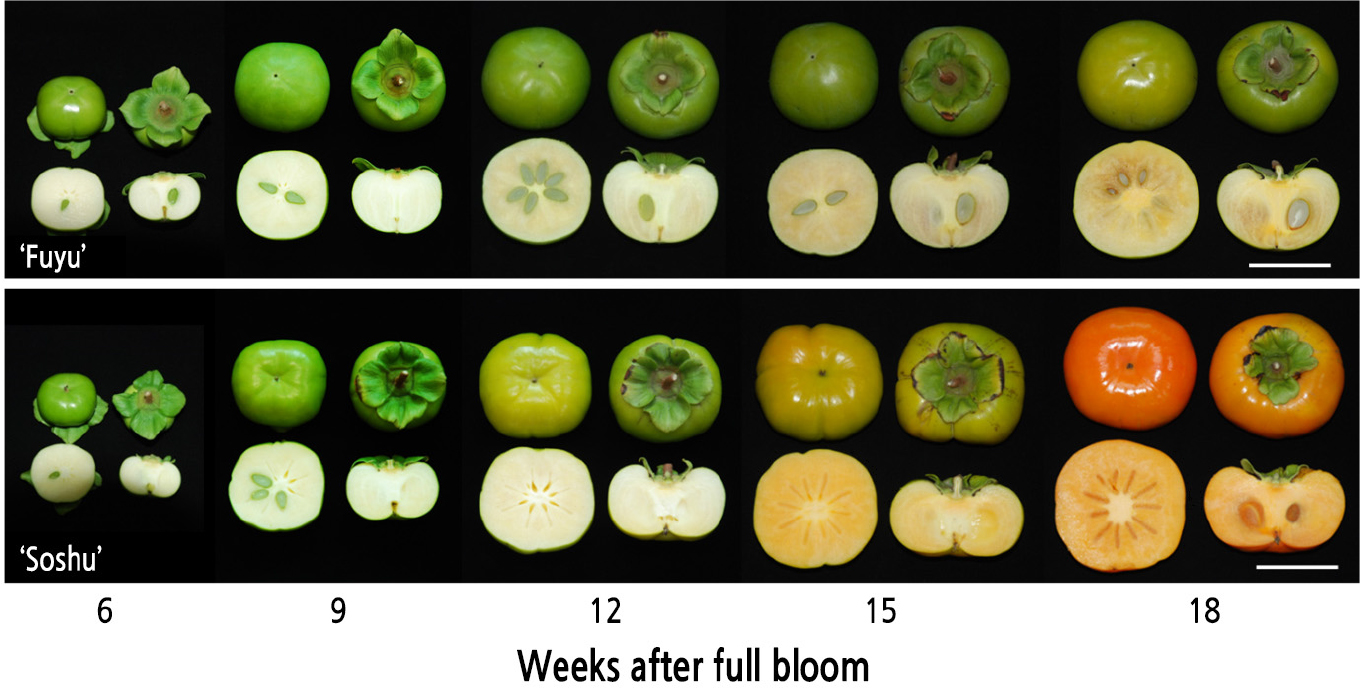
단감 과실 발육 단계별 색도 변화
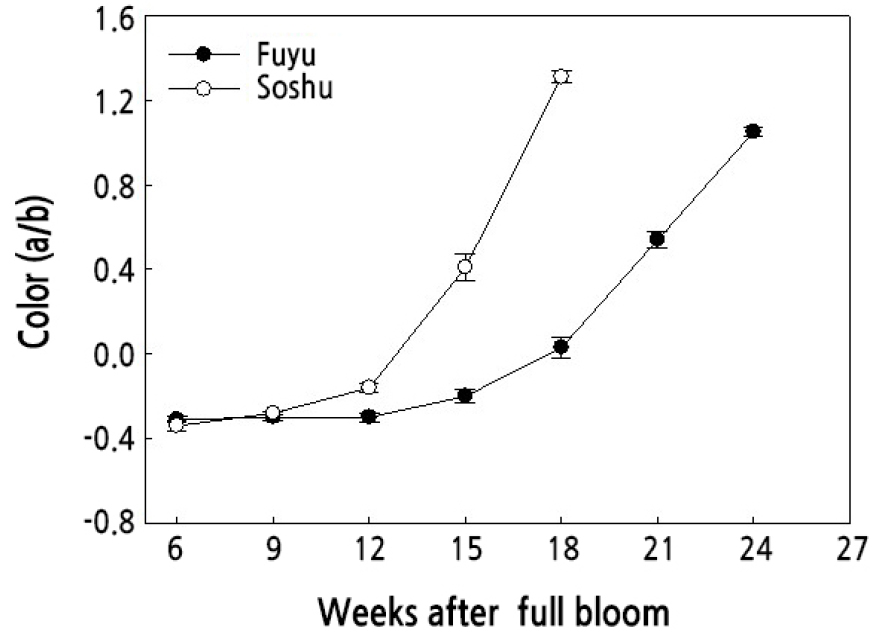
품종간 과실 성숙단계별 과색의 변화를 조사하였다(Fig. 1). 감의 과색은 주로 카로티노이드에 의한 것으로, ‘부유’와 ‘조추’ 품종 과피의 Hunter value a/b를 조사한 결과는 Fig. 2와 같다. ‘부유’ 과피의 a/b 값은 개화 후 12주까지는 거의 일정하다가 개화 후 12주 이후에 증가하기 시작하여 개화 후 15주부터 빠르게 증가하였다. ‘조추’ 과피의 a/b 값은 개화 후 9주까지 변화를 보이지 않다가 개화 후 9주 이후에 증가하기 시작해서 개화 후 12주부터 급속하게 증가하여 전반적으로 ‘부유’에 비해 ‘조추’에서 과피의 착색이 빠르게 진행되었으며, ‘조추’의 조사 종료 시점인 개화 후 18주에는 두 품종 간 과피의 a/b 값 차이가 현저하게 드러났다.
단감 과실 발육 단계별 유리당 함량 변화
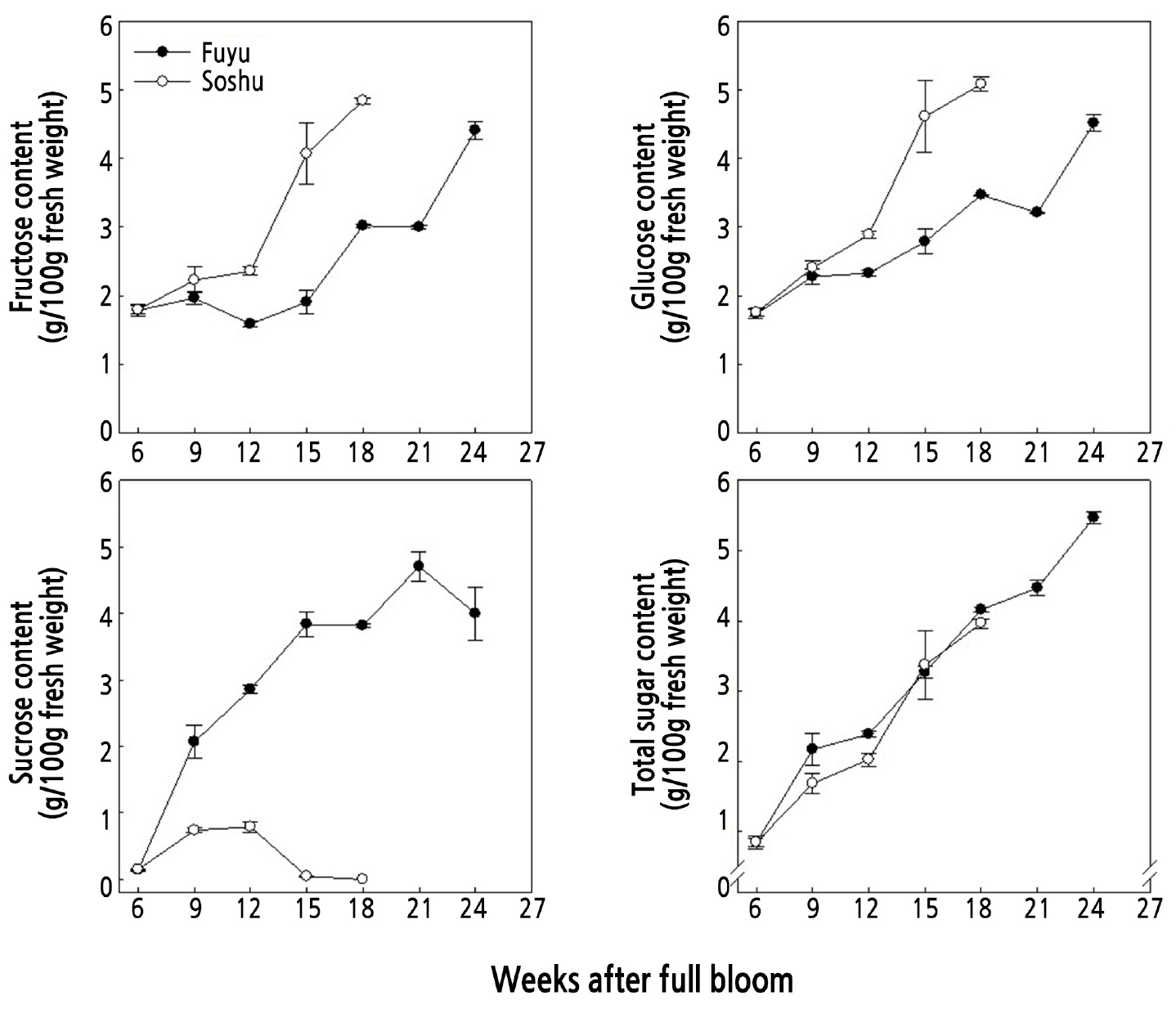
과실 내에 존재하는 주요 6탄당인 과당(fructose)과 포도당(glucose)의 함량은 ‘부유’는 개화 후 15주 이후에, ‘조추’는 개화 후 12주 이후에 급격히 증가하였다(Fig. 3). ‘부유’의 과실 내 서당(sucrose) 함량은 개화 후 6주부터 15주까지 지속적으로 급격히 증가하다가 개화 후 21주 이후에 감소되기 시작하여 조사 종료 시점에 100g FW(fresh weight) 당 약 4.0g 이었다. 반면 ‘조추’의 서당 함량은 개화 후 6주부터 12주까지 증가하다가 개화 후 12주 이후에 현저하게 감소하여 개화 후 15주의 서당 함량은 100g FW 당 약 0.05g 이었고, 조사 종료 시점인 개화 후 18주에는 서당이 거의 검출되지 않았다.
결과적으로 ‘조추’의 서당 가수분해가 ‘부유’보다 이른 시기에 진행되었는데, 잎에서 합성된 서당은 과실 내 과육으로 전류되어 효소에 의해 포도당과 과당으로 가수분해됨으로써 성숙한 과실의 서당 함량이 낮게 측정되므로 (Kakiuchi and Itoo, 1968), 서당 함량의 감소가 시작되는 시점을 과실 성숙의 개시점으로 볼 수 있고, 본 연구결과로 미루어 보면 ‘조추’ 품종의 과실 성숙이 ‘부유’보다 9주 가량 빨리 진행되는 것으로 나타났다.
과실 내 포도당, 과당, 서당을 포함하는 총당 함량을 조사한 결과 ‘부유’는 개화 후 6주에서 9주 사이에 급격히 증가했다가, 이후 3주간 완만하게 증가한 후 개화 후 12주 이후에 다시 현저하게 증가하여 조사 종료 시점인 개화 후 24주에는 ‘부유’ 과실 내 총당 함량이 100g FW 당 약 12.94g에 달했다. ‘조추’도 개화 후 6주부터 9주까지는 급격히 증가한 후 이후 개화 후 12주까지는 증가폭이 다소 둔화되었다가, 개화 후 12주 이후에 크게 증가하여 조사 종료 시점인 개화 후 18주에 과실 내 총당 함량이 100g FW 당 약 9.93g 이었다.
전사체 de novo Assembly
어셈블리된 전사체(NCBI accession no: PRJNA688576)를 k-mer별로 비교해서 total length와 average length 등을 고려하여 편차가 적은 k-mer, 59를 최적의 k-mer로 선정했고, k-mer = 59로 통합한 결과 총 104,574개의 전사체가(length > 200bp) 확보되었다(Table 1). 이후 매우 유사한 redundant transcripts를 제거하여 34,292개의 unigene 전사체를 확보하였다. Unigene 의 총 길이는 34,760,704bp, 평균 길이는 1,013bp, 최대 길이는 16,898bp였고, N50에서의 길이는 1,759bp였다(Table 1)(SM 1). 각 품종별 생산된 read 수와 read별 평균 길이는 Table 2와 같다.
Table 1.
Statistics of the de novo assembly for transcripts from fruit tissue of late-ripening persimmon cultivar ‘Fuyu’ and early-ripening ‘Soshu’
Table 2.
Statistics of the trimmed transcriptome sequencing data for fruit tissue of late-ripening persimmon cultivar ‘Fuyu’ and early-ripening ‘Soshu’
| Sample IDz | Num. of reads | Avg. length (bp) | Total length (bp) |
| B7 | 13,788,623 | 90.27 | 1,244,669,181 |
| 13,788,623 | 86.97 | 1,199,225,414 | |
| B8 | 9,948,572 | 84.78 | 843,434,394 |
| 9,948,572 | 80.45 | 800,370,328 | |
| B9 | 13,697,259 | 89.83 | 1,230,362,517 |
| 13,697,259 | 78.17 | 1,070,679,627 | |
| B10 | 10,581,409 | 84.32 | 892,199,164 |
| 10,581,409 | 79.62 | 842,440,181 | |
| J7 | 13,870,478 | 89.48 | 1,241,097,418 |
| 13,870,478 | 88.51 | 1,227,677,655 | |
| J8 | 11,273,123 | 89.97 | 1,014,294,452 |
| 11,273,123 | 87.69 | 988,572,186 | |
| J9 | 9,353,682 | 88.91 | 831,676,462 |
| 9,353,682 | 86.30 | 807,262,234 | |
| J10 | 9,553,720 | 89.25 | 852,710,020 |
| 9,553,720 | 86.12 | 822,780,309 | |
| 8ea | 184,133,732 | 86.40 | 15,909,451,542 |
전사체 Functional Annotation
총 34,292개 unigene을 Phytozome, Uniprotkb viridiplantae, Interproscan, KOG, KEGG 및 GO에 BLASTX 하였다. 그 결과, 19,428개(56.65%)의 unigene이 Phytozome database에서 42개 식물종 유전자와 sequence similarity를 나타내었다. 가장 많이 annotation된 식물종은 Vitis vinifera(23.72%)였고, Theobroma cacao, Manihot esculenta, Prunus persica , Solanum tuberosum , Populus trichocarpa가 그 뒤를 이었다(SM 2). 단감 전사체가 Vitis vinifera에 가장 많이 매칭된 이유는 크게 두 가지로 설명할 수 있는데 첫째, 단감과 Vitis vinifera 간 서열 유사성을 들 수 있고, 두 번째로는 database 내에 존재하는 Vitis vinifera의 시퀀스 수가 다른 식물종에 비해 상대적으로 많았기 때문으로 볼 수 있다(Gupta et al., 2015). 또한 본 결과는 Jung et al.(2017)이 ‘부유’ 과피를 재료로 Phytozome database에 annotation한 결과와 유사하기는 하나, 각 식물종에 매칭된 유전자수의 비율이 상이하고, Populus trichocarpa보다 Solanum tuberosum의 유전자와 annotation된 것이 더 많은 점 등이 달랐는데, 이는 표준유전체(genome) 서열을 이용한 annotation이 아니라 각각의 RNA-seq을 통해 생산된 데이터로 assembly한 gene set을 annotation에 이용했기 때문인 것으로 판단된다.
본 연구에서 확보한 unigene의 기능적 annotation을 위해 GO 분석한 결과 총 16,888개 unigene(49.24%)이 적어도 하나의 GO term에 해당되었는데, BP 카테고리에서는 metabolic process(9,595 unigenes)와 developmental processv(3,240 unigenes)가 가장 큰 하위 범주였고, CC 카테고리에는 다수의 unigene이 cell, organelle에 분류되었다. MF 카테고리에서는 catalytic activity(6,671 unigenes)와 transporter activity(926 unigenes)가 많은 비율을 차지했으며(SM 3), 애기장대 및 블루베리(Mitsuda and Ohme-Takagi, 2009; Gupta et al., 2015)와 유사하게 많은 유전자가 TF로 annotation되었는데, 이 시퀀스들은 과실 발육 및 성숙 단계에서 발현되는 관련 유전자들의 발현을 조절하는 것으로 추측된다.
26개 KOG 카테고리에 17,511개 unigene이 해당되었다. 이러한 카테고리 중에서 general function prediction only 클러스터는 가장 큰 그룹이었고(4,273: 24.40%), 그 다음이 signal transduction mechanisms(2,057: 11.74%), nuclear structure (1,754: 10.01%)이었다. 17,511개 unigenes의 17.84%(3,124개)가 signal transduction(group T) 혹은 transcription factors(group K)와 homology를 가졌는데(SM 3), 이는 토마토, 베이베리(Alba et al., 2005; Feng et al., 2012)의 경우보다 높은 수치였고, 단감 ‘부유’를 재료로 한 Jung et al.(2017)의 결과와는 유사하였다.
총 34,292개의 annotation된 시퀀스 중에서 6,288개의 unigene이 KEGG database에 매핑되었다. 매핑된 카테고리 중에서 가장 큰 것은 metabolism이었으며 carbohydrate metabolism(627 unigenes), amino acid metabolism(378 unigenes), lipid metabolism(301 unigenes), energy metabolism(238 unigenes) 등을 포함했다(SM 3). Genetic Information Processing pathway도 다수의 unigene을 포함했는데 translation(717 unigenes), transcription(350 unigenes), replication and repair (171 unigenes), folding, sorting and degradation(625 unigenes)이었다. Cellular Processes, Environmental Information Processing 및 Organismal Systems의 주요 카테고리는 각기 348, 636 및 833개의 unigene을 포함했다. 가장 대표적인 pathway는 탄수화물 대사(627 unigenes: 9.97%), 아미노산 대사(378 unigenes: 6.01%) 및 지질 대사(301 unigenes: 4.78%)였다.
과실의 발육은 전분 및 가용성 당의 과실 내 축적과 관련이 있고, 탄수화물 대사에 관한 유전자 발현 패턴을 조사하면 발육에 따른 과실특성의 형성에 대한 이해도를 높일 수 있는데(Xie et al., 2013), 본 연구에서 탄수화물 대사에 관련된 유전자들(627개)은 12개의 metabolic pathway에 분류되었고, 그 중 가장 많은 unigene을 포함한 것은 starch and sucrose metabolism(144개)과 Glycolysis/Gluconeogenesis(121개)였다. 이는 배(Pyrus bretschneideri Rehd)를 재료로 한 연구결과(Xie et al., 2013)와 유사하였으며, 이러한 annotation 결과는 단감의 생장과 발육에 관련된 specific process, pathway 및 기능 연구에 중요한 정보가 된다.
DEGs 분석 및 Clustering
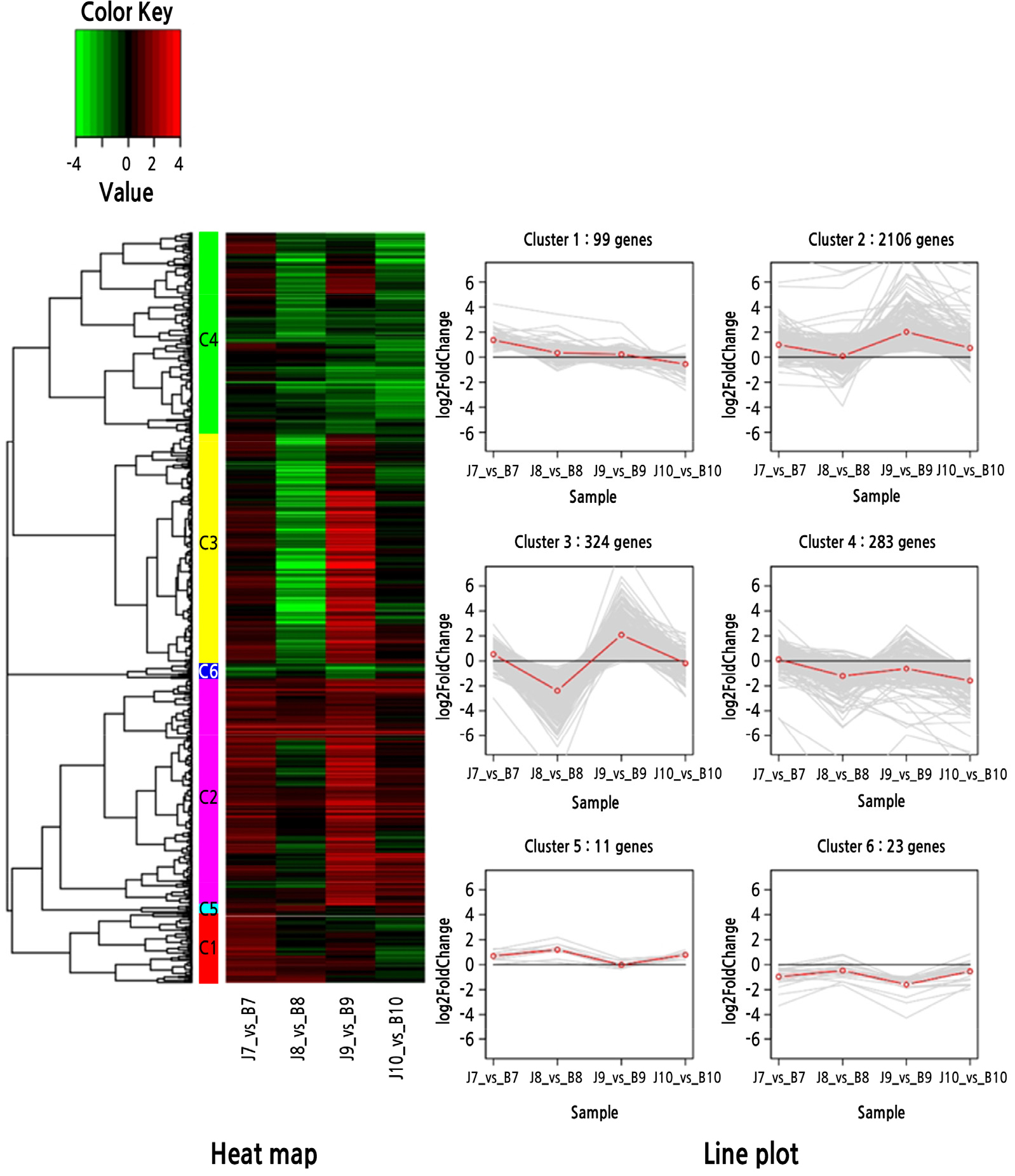
DEGs set별 발현량을 비교 분석한 결과, 동일 품종내 과실 발육 단계 간 분석인 set Ⅰ의 경우 ‘부유’는 3가지 비교조합 중에서 개화 후 18주에 대조구(개화 후 9주)에 비해 up-regulation된 유전자의 개수(1,886개)가 가장 많았고(SM 4)., 과실 발육 단계별 ‘부유’, ‘조추’ 간 비교인 set Ⅱ에서는 ‘조추’는 ‘부유’보다 3주가 이른 개화 후 15주에 대조구에 비해 up-regulation된 유전자의 개수(1,435개)가 가장 많았다(SM 5).
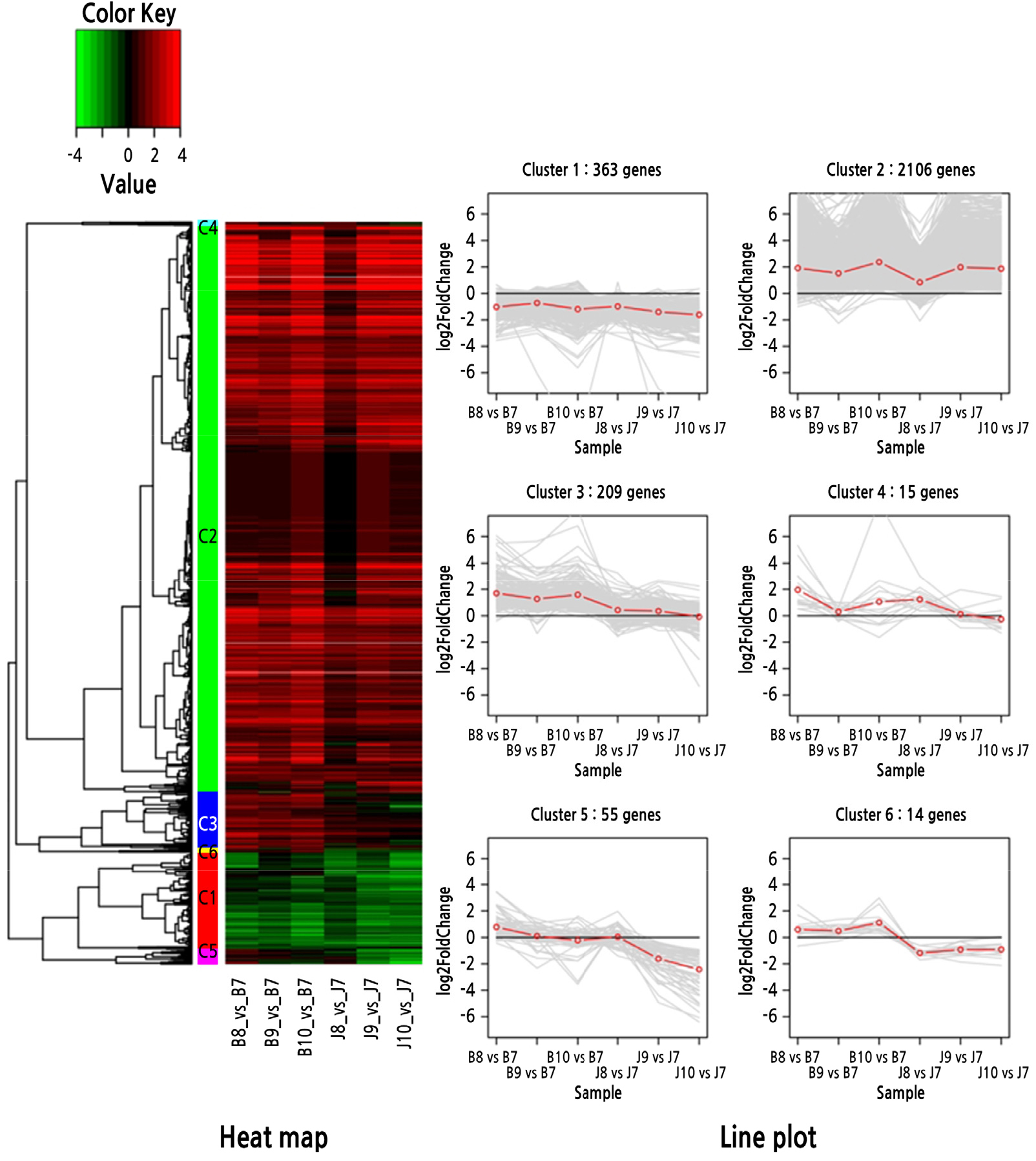
Clustering 분석을 위해 DEGs set별로 시간의 흐름에 따라서 같은 패턴으로 변화하는 유전자끼리 하나로 clustering한 결과 각각 6개의 cluster가 형성되었다. 품종별 과실 발육 단계간 DEGs를 clustering 분석한 set Ⅰ에서는, 6개 cluster 중에서 cluster 2는 DEGs 수가 2,106개로 가장 많으면서도, 나머지 cluster와 다르게 발현이 계속 높은 것으로 나타나, 성숙에 관련이 있는 유전자들일 것으로 예측되었고(Fig. 4), 과실 발육 단계별 ‘부유’, ‘조추’ 간 비교인 set Ⅱ에서는 cluster 2의 319개 유전자는 ‘부유’에 비해 ‘조추’에서 발현이 계속 up인 상태이고, 반대로 cluster 4의 283개 유전자는 ‘조추’에서 발현이 계속 down인 상태를 나타내었는데, ‘조추’가 ‘부유’에 비해 성숙이 빠른 품종이므로 cluster 2는 성숙을 촉진하는 유전자들의 up-regulation을, cluster 4는 성숙을 방해하는 유전자들의 down-regulation을 보여주는 것으로 판단되었다(Fig. 5).
Clustering set Ⅰ에 대한 GO 분석을 수행한 결과, cluster 2는 주로 transport(424), regulation of metabolic process(267), carbohydrate metabolic process(237), multicellular organismal development(215)에 관한 기능을 하며, intracellular membrane-bounded organelle(1,279)과 cytoplasm (1,055)의 위치에서, nucleic acid binding(312)과 nucleotide binding(174)에 관련된 분자적 기능을 가진 유전자가 많았으며, 이러한 결과를 KEGG 분석으로 확인했을 때 cluster 2는 biosynthesis of other secondary metabolites(554), carbohydrate metabolism(245), translation(212)에 관련된 pathway의 유전자들이 많은 것으로 나타났다(SM 6).
Clustering set Ⅱ에 대한 GO 분석 결과, cluster 2는 주로 transport(71)와 carbohydrate metabolic process(60)에 관련된 유전자가, cluster 4는 cell communication(64)과 transport(54)에 관련된 유전자가 많았으며, cluster 2 와 4 모두 주로 작동하는 위치가 cytoplasm이었으며, nucleotide binding과 nucleic acid binding에 관련된 기능을 하는 것이 많았는데, KEGG 분석을 통해 cluster 2는 biosynthesis of other secondary metabolites(165), carbohydrate metabolism(84)에 관련된 pathway의 유전자가, cluster 4는 biosynthesis of other secondary metabolites(55), environmental adaptation(42)에 관련된 pathway에 속하는 유전자가 많음을 알 수 있었다(SM 7).
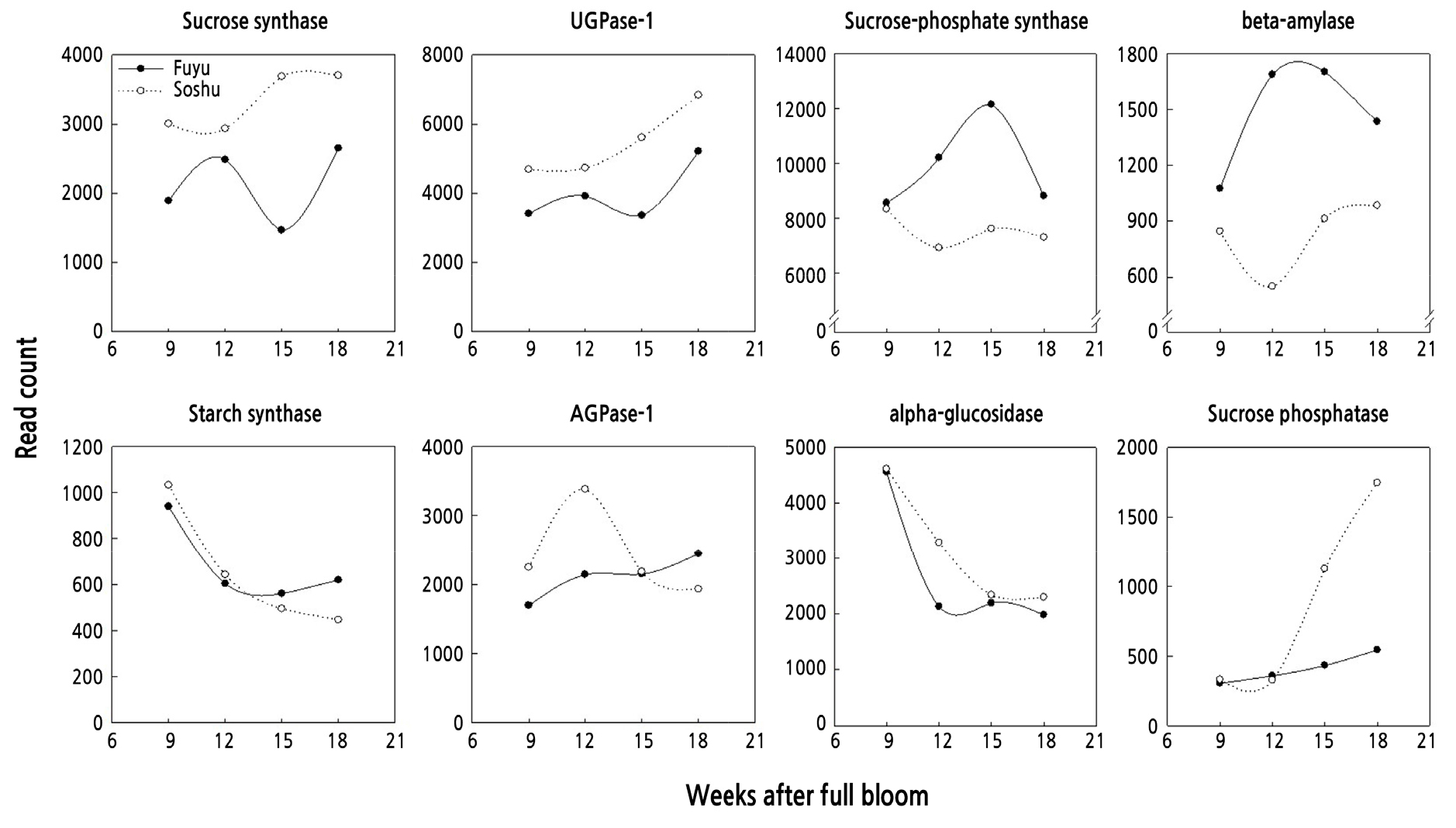
탄수화물 대사 관련 유전자 발현 양상
탄수화물 대사 관련 유전자로 Sucrose synthase 등 8개의 유전자에 대한 발현양상을 비교분석 하였다(Fig. 6). 이들 유전자 중 특히 UGPase는 주로 sucrose phosphate synthetase에 uridine 5'-diphosphoglucose(UDP-Glc)를 제공함으로써 서당 생합성에 관여하는데(Huber et al., 2000), 본 연구에서 탐지된 UGPase-1의 발현 프로필을 분석한 결과 개화 후 15주에 ‘부유’의 발현값이 3,367인 반면, ‘조추’는 5,613으로서 ‘부유’를 대조구로 한 log2 fold change 값이 약 0.74였다(Fig. 6). 감은 품종과 발육 단계에 따라 총당 함량 중 서당이 차지하는 비율에 차이는 있으나 ‘부유’의 경우 대략 30% 전후였다가 성숙기 이후에 그 비율이 크게 감소하게 되는데(Senter et al., 1991), ‘부유’에 비해 ‘조추’는 모든 단계에서 UGPase-1의 발현량이 많으면서도 특히, 개화 후 15주에는 발현량 차이가 크게 증가하였으므로, UGPase-1에 의한 서당 생합성 증가로 인해 과실 내 당 축적이 촉진되고 있음을 알 수 있었다.
또한 AGPase는 전분 생합성 경로의 첫 번째 효소로 glucose-1-phosphate와 ATP에서 ADP-glucose와 pyrophosphate를 생성하는 반응을 촉매 하는데(Ahn et al., 2009), ‘부유’는 유전자 발현값을 조사한 4단계에 걸쳐 AGPase-1의 발현이 지속적으로 증가하였으나, ‘조추’는 개화 후 12주를 기점으로 발현이 급격히 감소하였고, 이 시점에 ‘부유’, ‘조추’의 발현값은 각기 2,141과 3,382로서, 품종간 log2 fold change 값이 약 0.66이었다. 과실이 성숙되는 과정에는 전분 축적에서 당 축적으로의 이행이 필수적이므로(Janssen et al., 2008), AGPase-1의 발현이 개화 후 18주까지 지속적으로 증가했던 ‘부유’와 달리 ‘조추’는 개화 후 12주 이후 크게 감소 하였으므로, 전분 축적 감소와 이후의 당 축적 증가로 이어지는 과정이 만생종 ‘부유’에 비해 조생종 ‘조추’에서 이른 시기에 진행되고 있는 것이 확인되었다.
카로티노이드 생합성 관련 유전자 발현 양상
카로티노이드 생합성 관련 유전자로 GGPS 등 8개의 유전자에 대한 발현양상을 비교분석 하였다(Fig. 7). 특히 CRTISO는 고등식물에서 카로티노이드 생합성 경로의 isomerase로서 작용하여 poly-cis-carotenes를 all-trans 배열로 이성질화시키는 역할을 하고(Park et al., 2002), PDS는 구조적, 기능적으로 ZDS와 유사하며 phytoene을 zeta-carotene을 경유하여 lycopene으로 전환시키는 작용을 한다.
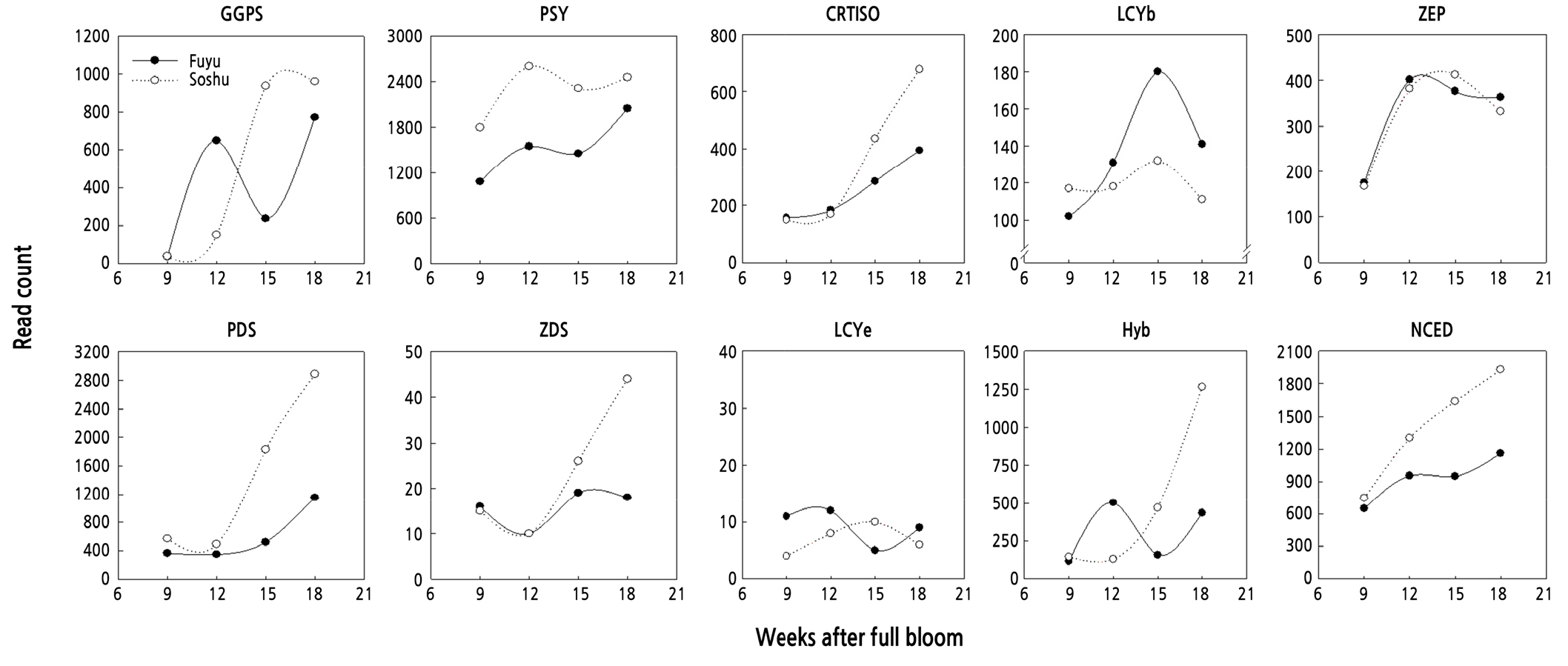
Fig. 7.
Expression pattern of the carotenoid biosynthesis genes at different stages of fruit development of the late-ripening persimmon cultivar ‘Fuyu’ and early-ripening ‘Soshu’. GGPS: Geranylgeranyl pyrophosphate synthase. PSY: phytoene synthase, CRTISO: carotenoid isomerase, LCYb: Lycopene beta-cyclase, ZEP: Zeaxanthin epoxidase, PDS: phytoene desaturase, ZDS: ζ-carotene desaturase, LCYe: Lycopene epsilon cyclase, Hyb: beta-hydroxylase, NCED: 9-cis-epoxycarotenoid dioxygenase.
CRTISO와 PDS 유전자의 발현량 변화를 분석한 결과 ‘부유’, ‘조추’ 과피색의 Hunter value a/b 그래프와 변화 패턴이 동일하였고, PDS는 카로티노이드 생합성 경로 유전자 중 발현량이 가장 많았다(Fig. 7). 두 품종 간 과피색 차이가 커지기 시작하는 개화 후 15주 시점의 CRTISO 발현값은 ‘부유’ 285, ‘조추’ 435로써 ‘부유’를 대조로 한 log2 fold change가 0.61이었다가, 이후 18주에 각기 발현값이 394, 678이 되어 log2 fold change가 0.78로 증가하였다. PDS의 log2 fold change는 15주에 1.82에서 18주에 1.33으로 다소 감소하긴 하였으나, 과피색의 Hunter value a/b와 유전자 발현량 변화를 통합한 그래프를 토대로 PDS는 CRTISO와 함께 ‘조추’ 과실의 착색을 촉진시키는 유전자임을 알 수 있었다.
NCED는 고등식물의 카로티노이드 생합성 경로에서 생성된 violaxanthin(9-cis-epoxycarotenoid)을 쪼개어 ABA의 C15 전구체인 xanthoxin을 생성하는 ABA 생합성의 핵심 효소이고(Iuchi et al., 2001), ABA는 과실 내 당 축적과 climacteric 및 non-climacteric type의 과실 성숙을 촉진하는 역할을 한다(Pan et al., 2005). 본 연구에서 NCED 유전자의 발현은 ‘부유’, ‘조추’ 모두 발육이 진행됨에 따라 증가하였고, ‘조추’에서는 개화 후 9주 이후에 발현이 급격히 증가하였다. 특히 개화 후 15주의 발현값은 ‘부유’, ‘조추’가 각각 949, 1638로써 품종 간 log2 fold change가 약 0.79에 달하여 ‘부유’에 비해 ‘조추’에서 NCED 유전자가 많이 발현되고 있음을 확인하였다.
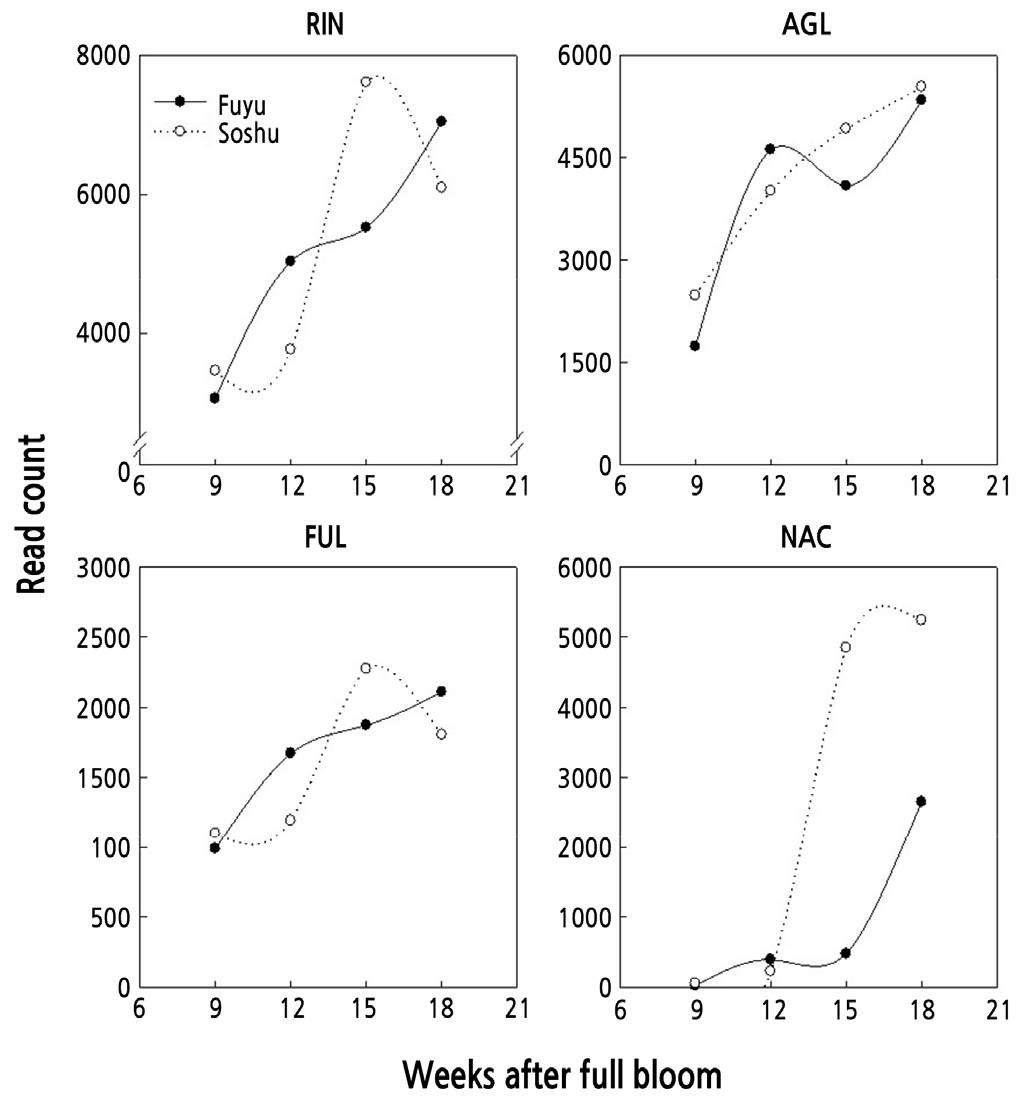
성숙 조절 전사인자 발현 양상
성숙을 조절하는 것으로 알려진 MADS-box 전사인자(transcription factor, TF)로 ripening inhibitor(RIN), AGAMOUS- LIKE(AGL), FRUITFULL(FUL)이 있으며, RIN은 ethylene-dependent regulation의 upstream에 존재하고 SEPALLATA- type MADS-box TF로 작용한다(ITO, 2016). 한편, NAM, ATAF, and CUC(NAC)는 선행연구에서 바나나와 복숭아의 성숙 조절과 연관이 있는 것으로 보고된 전사인자이다(Shan et al., 2012; Pirona et al., 2013).
RIN, AGL, FUL, NAC의 발현량을 분석한 결과, RIN과 FUL의 경우 단계별 발현 패턴이 매우 유사하였고, 유전자의 발현값 peak가 ‘부유’보다 ‘조추’에서 3주 빨랐으며, 개화 후 15주의 ‘부유’ 대비 ‘조추’의 log2 fold change는 각기 0.46, 0.28이었다(Fig. 8). AGL은 개화 후 12주에서 15주 사이 발현이 감소한 ‘부유’와 달리 ‘조추’의 발현은 조사기간 중 지속적으로 증가하였고, 개화 후 15주에서 18주 사이 ‘조추’에서 발현이 감소하는 RIN, FUL과도 다른 양상을 나타내었으며, ‘조추’에서 개화 후 15주에 ‘부유’에 비해 발현량이 많았다.
RIN, AGL, FUL은 선행연구에서 ACS의 promoter에 결합하여 ACS의 전사를 촉진하고, 이로 인한 에틸렌 생합성 증가를 통해 과실의 성숙이 촉진되므로, 본 연구에서 RIN, AGL, FUL의 발현은 개화 후 15주에 ‘부유’에 비해 ‘조추’에서 발현량이 많았으므로, ‘조추’에서 이들 전사인자에 의한 ACS 전사 촉진으로 인해 에틸렌 생합성 증가와 성숙 촉진이 진행되고 있는 것으로 추정되었다.
NAC는 ‘조추’에서 개화 후 15주에 발현이 급격히 증가하여 9주 대비 log2 fold change가 6.63이면서, 동 시점의 ‘부유’ 대비 log2 fold change는 3.37에 달하여 ‘부유’보다 ‘조추’에서 NAC 유전자의 발현이 월등히 많은 것을 알 수 있었다. 단감에서 NAC의 기능은 아직 밝혀지지 않았으나, NAC가 동일한 climacteric type인 바나나와 복숭아의 성숙 조절과 연관이 있으므로 단감의 성숙 조절에도 기능을 가질 것으로 예측되었다.
본 연구에서 밝혀진 동일 품종별 과실 발육 단계간, 그리고 동일 과실 발육 단계별 ‘부유’와 ‘조추’ 간 차등적으로 발현된 유전자들은 단감 과실 숙기조절의 유전적 메커니즘을 이해하는데 유용한 자료가 될 것이다. 특히 발현 양상에 주요한 차이를 보이는 것으로 판단되는 탄수화물 대사, 카로테노이드 생합성 및 성숙 조절인자 유전자들은 향후 과실 조숙형질 선발용 분자마커 개발을 위해 활용될 수 있을 것으로 사료된다.