서 언
재료 및 방법
실험재료 및 차나무 재배조건
Primer 제작
Molecular works (RNA extract, Reverse Transcription & PCR conditions)
Real-time PCR 반응 조건
Amino acid 분석
통계분석
결과 및 고찰
테아닌 합성유전자 선별 마커 선발
Real-time PCR
아미노산 분석
서 언
차는 차나무(Camellia sinensis L.)의 잎을 다양한 방법으로 가공하여 음용하는 대표적인 기호 식품 중 하나이다. 차의 종류는 일반적으로 녹차, 반발효차, 발효차와 미생물 후발효차로 구분되며, 차의 품질은 찻잎의 채취시기, 제다시 발효방법과 발효강도에 따라 추출물의 성분과 맛의 차이를 가져오며, 품질을 결정짓는 요인은 매우 다양하다. 또한, 차나무 품종, 재배환경 및 재배기술 등에 따라 차의 품질이 달라질 수 있다.
차나무의 교배육종기간은 약 25년으로 정도가 소요된다고 알려져 있다(Liang and Feng 2000; Takeda 2000; Jeong and Park 2012). 일본은 1953년 선발육종을 통해 ‘야부기다’ 품종을 보급하였으며, 최근 6,000여점의 유전자원을 보유하고 있다. 또한, 중국은 18세기 중엽부터 품종육성을 시작하였으며 1990년대 초부터는 정부에 의해 진행되었다(Takeda 2000; Tanaka 2012). 그러나, 한국의 차나무 품종육성은 1990년대 초반에 시작하여 현재 18종의 품종이 등록되었고, 국가적 지원을 통한 차나무 육종사업은 2016년 농촌진흥청 국립원예특작과학원에서 차나무 육종연구를 시작했다(Service 2022). 또한, 국내에 재배되고 있는 차나무 품종은 야부기다 및 후슌 등의 일본 품종이 제주도에서 우점/재배되고 있으며, 전남, 경남 및 전북 등 타 지역은 야생종화된 재래종 차나무가 재배되고 있는 실정이다. 차나무 재래종은 개체간 생장시기가 일정하지 않아 기계화가 어려울 뿐만 아니라, 생산된 찻잎의 품질이 동일하지 않은 단점을 가지고 있다(Yoon et al. 2016). 이에 농촌진흥청, 전라남도 농업기술원, 제주특별자치도 농업기술원등에서 국내육성 품종을 개발하였으나, 국내육성 품종의 보급률이 현저히 낮을 뿐만 아니라 2017년 8월 17일 나고야의정서 국내 발효에 따라 국내품종의 개발이 더욱 절실해진 상황이다. 우수한 품질의 차 생산은 우수한 형질을 가진 차나무 품종을 육종할 필요가 있다. 차나무 품종 선발은 다수성, 내병성, 기상에 대한 내재해성(동해, 가뭄 등) 및 우수한 품질(향, 수색, 맛)등의 기준을 설정하여 각각의 특성에 맞는 차나무를 선발 및 육종하여야 한다(Kim et al. 2002; Lee et al. 2010). 일반적으로 차는 pigment, saponins, gallic acid, methoxy phenolic compound, poly- flavonoids을 다량 함유하고 있어 항균 작용, 혈중 콜레스테롤 감소 효과, 항산화 활성 등의 다양한 생리활성이 보고되었다(Lv et al. 2013; Tang et al. 2019). 또한, 차에 함유된 아미노산(glutamate, alanine, lysine, tyrosine, theanine 등)은 성분과 함량의 차이로 인해 맛에 영향을 주며 품질과 가격에 영향을 준다고 알려져 있다(Kim et al. 2022). 차에 함유되어 있는 아미노산 중 테아닌(theanine)은 차 품질에 큰 영향을 미치는 특별한 감칠맛 때문에 차의 품질 평가에서 중요한 지표로 자리매김하고 있으며, 정신적 이완효과(불안 및 스트레스 해소), 혈압강화 및 학습능력 향상 등의 효과가 보고되었다(Yamaguchi and Ninomiya 2000; Park et al. 2002; Mancini et al. 2017; Saeed et al. 2017; Lee et al. 2018). 따라서, 우수한 품질의 차를 생산하기 위해서는 테아닌 및 아미노산 함유량이 높은 차나무 품종의 개발이 매우 시급하다.
분자마커는 DNA & RNA 수준에서 두 개체간 차이를 구별해 낼 수 있는 선별시스템이다. 1970년대 다양한 분자마커가 개발되어 분류군간 식별, 유전자 빈도를 활용한 집단유전학 연구, 생물 종 다양성 연구, 생물체내 유전자의 특성과 기능을 규명하는 연구 등으로 적용분야가 확대되고 있다(Kim et al. 2014c; Huh 2015; Kim et al. 2021a; Yang et al. 2021b). 식물육종시 분자마커의 응용은 우수 형질을 가진 개체들을 효율적인 선발을 가능하게 하고, 순도 향상, 육종 연한 및 노동력 감소라는 큰 효율성을 줄 수 있다. 특히, 병충저항성, 기후에 대한 저항성과 특정형질을 가진 원예작물 품종 개발 분야에서 매우 유용하게 활용되고 있다(Kim et al. 2014b; Park et al. 2016; Kim et al. 2018; Kim et al. 2021b; Ni et al. 2023).
이에 본 연구는 차나무 육종시 유목 단계에서 테아닌 함량이 높은 개체를 조기에 선별할 수 있는 기술에 관한 연구를 수행하였으며, 대량신속검색기술(High Throughput Screening system, HTS system)에 활용할 수 있는 분자마커를 개발하고자 하였다.
재료 및 방법
실험재료 및 차나무 재배조건
차나무 품종은 전라남도 농업기술원에서 육성된 참녹과 보향으로부터 삽수를 채취하고, 2년생 삽목을 이용하였다. 2년생 삽목은 포트에 이식한 후, 4개월간 유리온실에서 안정기를 거쳤다. 차광실험은 검은색 셀로판지(dark 50%)와 불투명한 재질(검정색 polycarbonate, dark)로 만든 박스(암조건)로 2주간 차광하였으며, 2주간 배양한 후 잎을 채취하여 분석하였다.
Primer 제작
테아닌 합성관련유전자는 NCBI(www.ncbi.nlm.nih.gov)에서 조사하였으며, Camellia sinensis theanine synthetase mRNA(JN226569) 염기서열을 이용해 molecular probe를 제작하였다. Primer 설계는 24 mer, tm 60–67°C, GC content 40–60%, product size 200–400bp로 설정하여 primer3(ver 0.040) (https://bioinfo.ut.ee/primer3-0.4.0/)를 이용하여 primer를 제작하였다(Untergasser et al. 2012)(Table 1).
Molecular works (RNA extract, Reverse Transcription & PCR conditions)
RNA 분리를 위해서는 Plant RNA extraction kit(RNeasy Plant Mini Kit, Qiagen, Hilden, Germany)를 이용하였으며, 분리한 total RNA의 농도 및 순도는 Nanodrop ND-2000(Thermo Scientific, Waltham, MA, USA)를 이용하여 확인하였다. Reverse Transcription은 RT kit(RT kit(M-MLV, RNase H-), Biofact, Daejeon, Korea)를 이용하여 수행하였으며, 시료에서 분리한 total RNA 250ng에 random hexamer 50µM을 첨가한 후, 혼합된 mixture를 65°C, 5분간 열을 가한 후 ice에 옮겼다. 차갑게 식힌 mixture tube에 5X RT reaction buffer(include dNTP mix) 4µL, 8 mM DTT 1µL, RTase(200U/µL) 1µL, RNase Inhibitor(40U/µL) 1µL를 첨가한 후, 전체 용량이 20µL가 되게 하였다. 이 혼합물을 50°C에서 30분간 reverse transcription 반응 후 95°C에서 5분 동안 가열하여 reverse transcriptase의 활성을 없애고 RT과정을 마무리하였다. RT과정을 마친 cDNA를 주형으로 PCR 과정을 수행하였다. 디자인된 primer를 이용하여 50µL 안에 10 × PCR buffer, 20mM MgCl2, 40mM dNTP mixture, 각 primer(1µM), template DNA와 0.5U Taq polymerase를 첨가하여 PCR mixture를 만들었다. 반응조건은 94°C에서 5min 동안 DNA를 pre-denaturation 시켜, 94°C에서 45sec denaturation, 62°C에서 45sec annealing, 72°C에서 45sec extension하고 72°C에서 5min 동안 final extension을 수행하였다. PCR 증폭산물을 1.5% agarose gel에 loading 한 후 band를 잘라, HiGeneTM Gel & PCR Purification System(Biofact, Daejeon, Korea)을 이용하여 정제하였다. 정제된 PCR product는 All-in-one vector(Biofact, Daejeon, Korea)에 ligation하고, 이 ligation mixture를 E. coli DH5α(RBC, Taipei, Taiwan)에 넣은 후 LB+kanamycin(50µg/mL) 배지에서 키워 생성된 colony의 염기서열을 확인하였다. 삽입된 16S rRNA 유전자의 염기서열은 3730xl DNA Analyzer(Thermo Scientific)를 이용하였으며, M13-20F and M13-20R(All in one Vector Systems manual) primer를 이용하여 결정하였다. 결정된 염기서열은 ClusterX를 이용하여 유전자간의 상동성을 분석하고, NCBI(www.ncbi.nlm.nih.gov)의 GenBank database를 이용하여 BLAST search를 통해 확인하였다(Kim et al. 2020).
Real-time PCR 반응 조건
염기서열이 확인된 plasmid DNA는 real-time PCR을 이용하여 melting curve(Bio-Rad) 분석 후 정량분석을 위한 표준 유전자(artificial standard clone)로 사용하였다. 정량 PCR을 위해 CFX96 TouchTM Real-Time PCR Detection System (Bio-Rad)과 iTaqTM SYBR® Green Supermix with ROX(Bio-Rad)를 이용하였다. Real-time PCR 조건은 gradient PCR을 통해 annealing 온도를 설정했으며, gradient PCR 조건은 95°C에서 15 min 동안 pre-denaturation 시켜 95°C에서 30 sec denaturation, 62–69°C에서 30 sec annealing, 72°C에서 30 sec extension 후 fluorescence를 측정하고 45cycles를 수행하였다. 그리고 final extension은 72°C에서 5min 동안 수행하였다. PCR product의 melting curve는 65°C부터 95°C까지 0.2°C씩 증가시키면서 fluorescence를 측정하여 분석하였다. 선별된 primer set를 통한 정량분석은 앞선 gradient PCR 분석을 통해 64.8°C로 annealing 온도를 설정하여 수행하였다. 정량을 위해 표준유전자를 serial dilutions 하여 real-time PCR을 수행하였으며, cDNA 농도는 1ng/µL을 분석하였다(Kim et al. 2012).
Amino acid 분석
테아닌 분석은 아미노산 분석을 위해 시료를 분쇄하여 분석에 이용하였다. 아미노산 추출은 0.25g을 측정하여 3차 증류수 10mL와 혼합한 후 autoclave를 이용하여 95°C에서 20분 동안 처리하였다. 추출액은 0.2µm Nylon syringe filter(Whatman, Buckinghamshire, United Kingdom) 여과한 후 HPLC(Agilent 1200 series, Agilent Technologies, Santa Clara, CA, USA)로 테아닌 함량을 분석하였다. 분석에 사용된 컬럼은 Zobrax Eclips AAA(4.6 × 150mm, 5µm, Agilent Technologies, Santa Clara, CA, USA)를 이용하였으며, 컬럼온도는 40°C로 설정하였다. 이동상은 phosphate buffer와 methanol: acetonitrile: DW(45: 45: 10)를 이용하였으며, 이동상의 유속은 2mL/min, running time은 26min으로 설정하여 분석하였다. 검출파장은 338nm와 262nm 파장에서 흡광도를 측정하여 분석하였다(Lee et al. 2018).
통계분석
실험은 최소 3반복으로 진행하였고, 각 시료들의 테아닌 유전자의 발현과 테아닌 함량을 분석하여 평균 ± 표준편차로 나타냈으며, 일원배치 분산분석(ANOVA)과 Duncan의 평균간 다중 비교를 이용하여 평균값을 비교하였다(Ahn et al. 2022).
결과 및 고찰
식물의 아미노산은 탄소와 산소, 수소를 광합성을 통해 탄수화물을 생성하며, 토양에서 얻은 질소와 결합하여 아미노산을 생성한다. 따라서 광합성은 식물의 아미노산 합성에 중요한 역할을 한다. 차의 아미노산 중 테아닌 함량 증가를 위해 차광을 통해 아미노산을 증가시킬 수 있으며, 차광을 하면 차광하지 않을 때보다 테아닌 함량이 약 2배 이상 증가된다고 보고되어 재배에 활용되고 있다(Sakai 1975; Okano et al. 1997; Song et al. 2013; Shim et al. 2019; Yang et al. 2021a). 그러나, 차광과 테아닌 합성과정의 유전적 관계는 명확하지 않은 실정이나, 차광으로 인한 테아닌 함량과 테아닌 합성효소의 단백질 발현이 함께 증가하는 것이 보고되어 차광과 테아닌 합성 유전자와의 밀접한 관계가 있다고 판단 할 수 있을 것이다(Deng et al. 2010). 이에, 본 연구에서는 차광으로 인한 테아닌 합성 유전자(tsa gene)의 발현과 테아닌 함량과의 상관관계를 분석하고 조기선별 마커로 활용 가능한지를 검증하고자 한다.
테아닌 합성유전자 선별 마커 선발
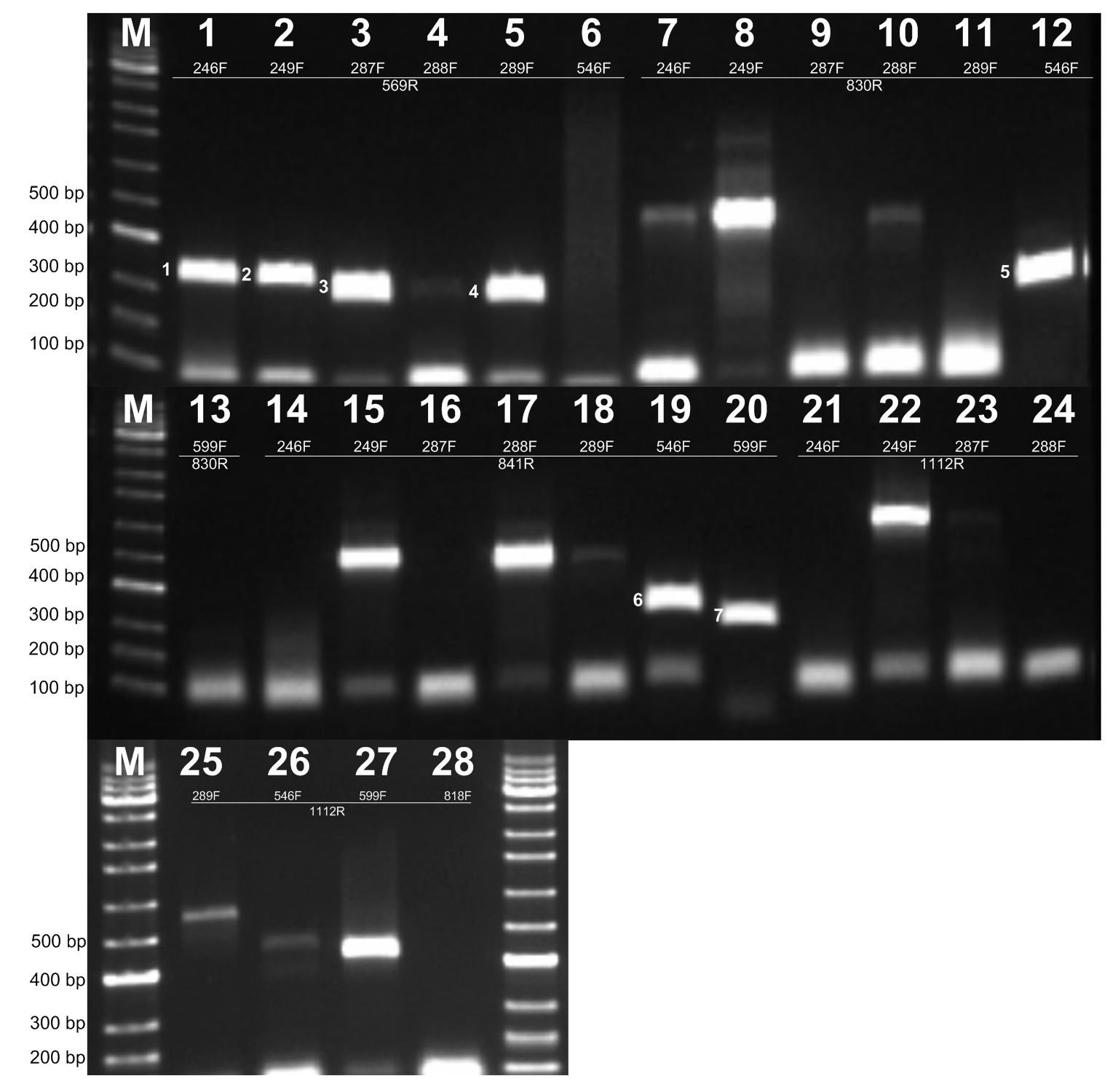
본 연구는 차나무 육종시 유목 단계에서 테아닌 함량이 높은 개체를 선별할 수 있는 기술을 개발하기 위해, 대량신속검색기술(High Throughput Screening system, HTS system)에 활용할 수 있는 테아닌 선별 분자마커를 개발하고자 하였다. 이를 위해 NCBI database(www.ncbi.nlm.nih.gov)와 primer3(ver 0.040)를 이용하여 primer를 설계하여 forward primer 8개, reverse primer 4개를 선별하여 제작하였다(Table 1). 제작된 primer set는 테아닌 합성유전자를 검출할 수 있는지를 확인하기 위해 PCR을 수행하였다. 선별기준은 PCR product size 200–400bp이며 하나의 밴드만을 생성하는 primer set로 설정하였으며, 이 조건을 만족한 primer set는 7개(TSA-246F/569R, TSA-249F/569R, TSA-287F/569R, TSA-289F/569R, TSA-546F/830R, TSA-546F/841R, TSA-599F/841R)로 확인되었다(Fig. 1). 선별한 7개의 PCR 밴드는 cloning을 수행하여 PCR 증폭산물의 크기와 염기서열을 확인하였다. 테아닌 합성유전자를 검출할 수 있는 primer 제작을 위해 사용한 Camellia sinensis theanine synthetase mRNA(JN226569) 염기서열과 cloning을 통해 확보한 염기서열의 상동성을 분석하였으며, 제작된 primer를 사용해 증폭된 PCR 산물은 Camellia sinensis theanine synthetase mRNA gene과 동일한 염기서열을 갖는 것을 확인하였다(자료 미제시).
Table 1.
Primer list for detection of the theanine synthetic gene in C. sinensis
| Primer name | primer sequence | Length (mer) | Tm (°C) | GC% | Start location |
| TSA-246F | GGACAAGCTCCTGGTGAAGATAGT | 24 | 62.18 | 50.00 | 246 |
| TSA-249F | CAAGCTCCTGGTGAAGATAGTGAA | 24 | 61.97 | 45.83 | 249 |
| TSA-287F | TCAAGCAATTTTTAAGGACCCATT | 24 | 62.05 | 33.33 | 287 |
| TSA-288F | CAAGCAATTTTTAAGGACCCATTC | 24 | 62.05 | 37.50 | 288 |
| TSA-289F | AAGCAATTTTTAAGGACCCATTCA | 24 | 62.05 | 33.33 | 289 |
| TSA-546F | ATTGGTGTTGACAAAGCTTATGGA | 24 | 61.91 | 37.50 | 546 |
| TSA-599Fz | TTGCCTCTATGCTGGTGTAAACAT | 24 | 62.04 | 41.67 | 599 |
| TSA-818F | TTACAGCACCAAGTCTACGAGGAG | 24 | 62.00 | 20.00 | 818 |
| TSA-569R | TCCATAAGCTTTGTCAACACCAAT | 24 | 61.91 | 37.50 | 569 |
| TSA-830R | CTTGGTGCTGTAATTGGTATGAGC | 24 | 62.04 | 45.83 | 830 |
| TSA-841Rz | CTCCTCGTAGACTTGGTGCTGTAA | 24 | 62.00 | 50.00 | 841 |
| TSA-1112R | CTCTGCAATCATGGAAGTAACCAC | 24 | 62.09 | 45.83 | 1,112 |
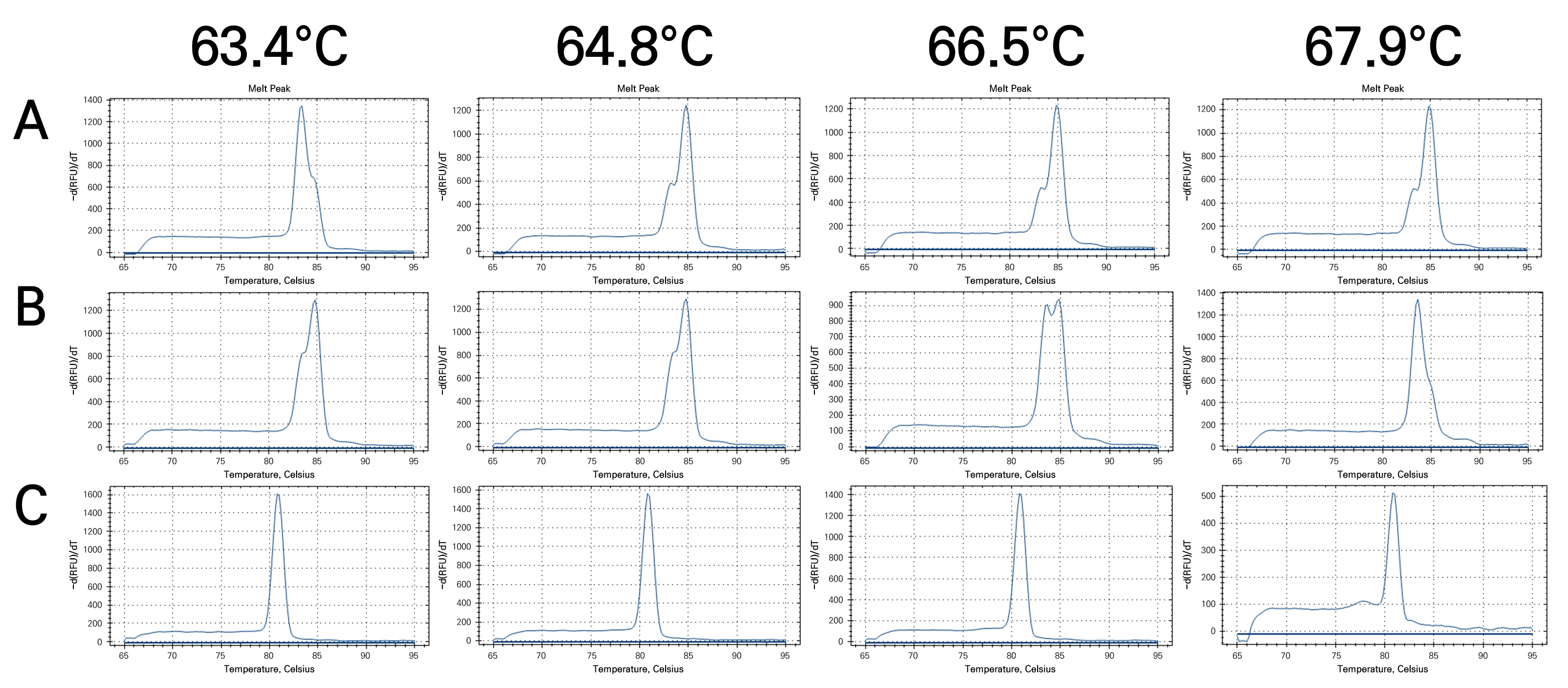
증폭된 7개의 테아닌 합성유전자 증폭산물은 cloning을 통해 얻은 테아닌 합성유전가가 삽입된 clone을 테아닌 합성 표준유전자로 활용할 수 있는지를 real-time PCR 기기를 이용해 gradient PCR을 수행한 후 melting curve 분석을 통해 확인하였다. 테아닌 합성유전자 clone은 melting curve를 분석한 결과 대부분의 테아닌 합성유전자 clone에서 2개 이상의 peak를 확인하였다(Fig. 2A and 2B). 그러나, TSA-599F/841R clone을 이용한 melting curve는 63.4°C부터 67.9°C까지 모든 annealing 온도조건에서 1개의 peak를 확인할 수 있었다(Fig. 2C). 이는 특정유전자를 검출하는 표준유전자와 primer set 선발에서 2개 이상의 melting curve peak는 측정시 검출 오류를 줄 수 있다고 알려져 있어, 표준유전자 및 선별 마커로서 사용하기 부적합하다고 판단했다(Kim et al. 2014a). 또한, melting curve peak의 높이는 64.8°C 까지는 1500정도 확인되고, 66.5°C에서는 1400, 67.9°C에서는 500으로 감소하는 것을 확인하여, 적정 annealing 온도는 64.8°C로 설정하였다. 테아닌 합성유전자의 염기서열 분석과 real-time PCR을 이용한 melting curve분석을 통해 표준유전자로는 TSA-599F/841R clone으로 선별하고, 프라이머는 TSA-F(TSA-599F)와 TSA-R(TSA-841R)로 결정하였다.
Real-time PCR
차광실험을 통해 참녹과 보향 잎을 채취하여 RNA를 추출하여 테아닌 합성유전자의 발현량은 real-time PCR을 통해 분석하였으며, 차광에 따른 광량에 따라 tsa gene 발현량은 유의한 차이를 나타내었다(Fig. 3 and Table 2). 차광하지 않은 차나무 참녹과 보향의 잎에서 테아닌 합성유전자의 발현은 각각 5.51 × 104 ± 5.44 × 103 copy·molecule/µL와 5.00 × 104 ± 3.85 × 103 copy·molecule/µL로 확인하였다. 그리고, 50% 차광한 차나무 참녹과 보향 잎에서 테아닌 합성유전자의 발현은 각각 8.31 × 104 ± 6.05 × 103 copy·molecule/µL와 8.10 × 104 ± 5.54 × 103 copy·molecule/µL로 확인되었다. 마지막으로 암조건에서 차나무 참녹과 보향 잎에서 테아닌 합성유전자의 발현은 각각 1.03 × 105 ± 9.94 × 103 copy·molecule/µL와 6.88 × 104 ± 4.10 × 103 copy·molecule/µL로 확인되었다.
Table 2.
The expression level of tsa gene and theanine content according to the light shading
차광하지 않은 차나무 참녹과 보향의 테아닌 합성유전자의 발현은 5.51 × 104 copy·molecule/µL와 5.00 × 104 copy·molecule/µL로 비슷한 수준으로 발현되며, 50% 차광한 조건에서 테아닌 합성유전자의 발현은 8.31 × 104 copy·molecule/µL와 8.10 × 104 copy·molecule/µL로 유사하게 발현된 것을 확인하여 품종이 다름에도 불구하고 테아닌 합성유전자의 발현정도가 비슷한 것을 확인했다. 그러나, 암조건에서 차나무 참녹과 보향의 테아닌 합성유전자의 발현은 각각 1.03 × 105 copy·molecule/µL와 6.88 × 104 copy·molecule/µL로 참녹의 테아닌 합성유전자가 보향에 비해 150% 높게 발현되는 것으로 분석되었다. 또한, 참녹의 테아닌 합성유전자의 발현은 차광하지 않은 차나무와 비교해 50% 차광시 150.9%, 암조건에서 186.8%로 유전자의 발현이 증가하는 것으로 분석되었고, 보향의 테아닌 합성유전자 발현은 차광하지 않은 차나무와 비교해 50% 차광시는 162.0%, 암조건에서 137.6%로 증가하는 것으로 분석되었다. 이를 통해 차광은 테아닌 합성유전자 발현을 유도하여 증가하는 것을 확인할 수 있었다. 일반적으로 차나무는 반음지성 식물로 내음성이 강해 숲속에서도 생육이 양호하고, 일사량을 감소시키면 테아닌의 함량과 엽록소가 증가된다고 알려져 있다. 테아닌 합성유전자의 발현은 차광으로 인해 증가되었다고 판단할 수 있다.
아미노산 분석
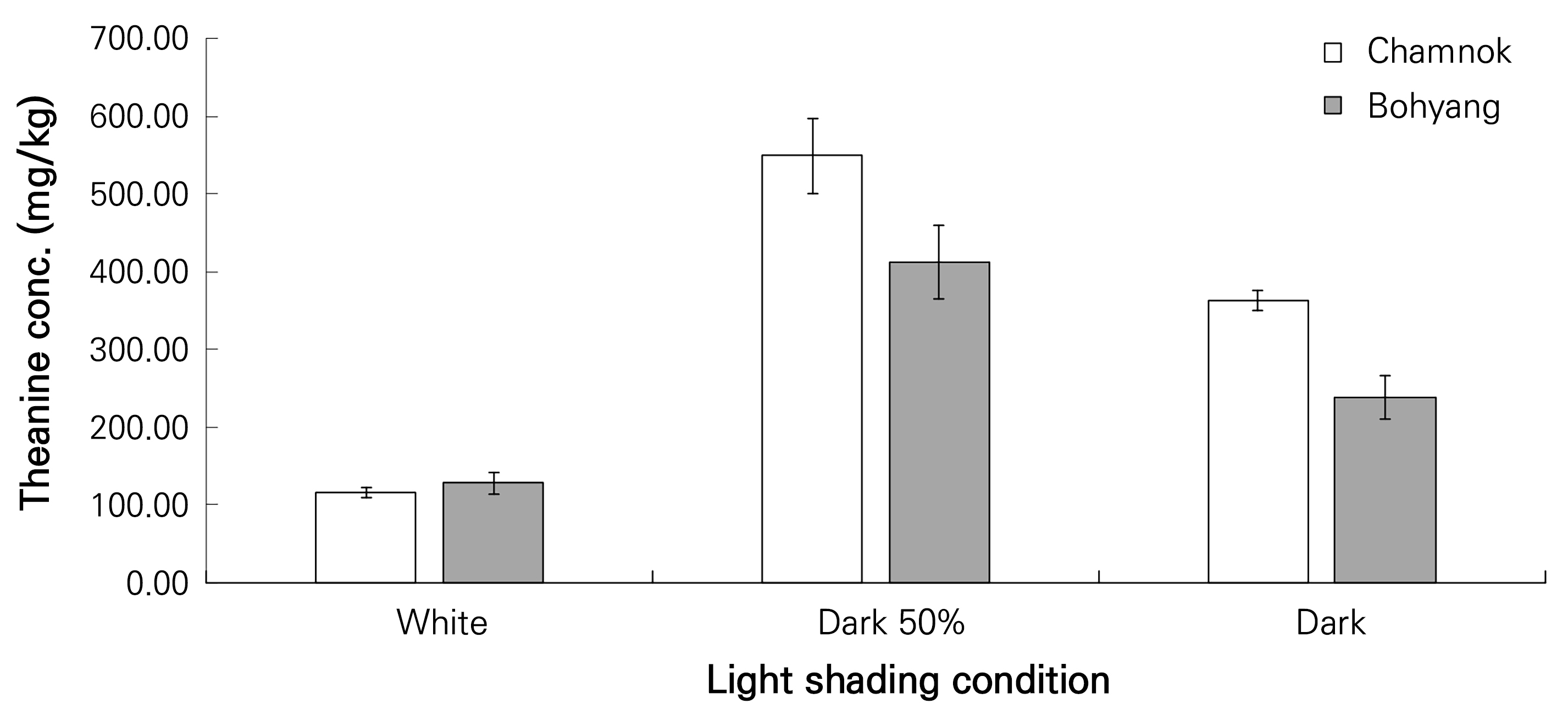
차는 테아닌과 데아플라빈(theaflavin)등의 아미노산함량이 높은 찻잎을 만드는 것이 중요하며, 이를 위해 수확 2주전 차광을 통해 찻잎의 아미노산 함량을 증대시킨다(Yang et al. 2021a). 테아닌 함량이 높은 차나무 자원의 식별 및 육종을 위한 분자마커 개발은 테아닌 대사 관련 유전자와 함량 사이의 상관 관계를 확인하는 것이 필수적이다. 이를 위해, 차광실험을 통해 차나무 잎의 테아닌 함량은 HPLC로 분석하고 테아닌 합성유전자의 발현정도를 비교하였다(Fig. 4). 또한, 테아닌 함량은 차광에 따른 광량에 따라 유의한 차이를 나타내었다(Table 2). 차광하지 않은 차나무 참녹과 보향 잎의 테아닌 함량은 각각 115.99 ± 6.09mg/kg과 127.75mg/kg인 것으로 확인하였다. 50% 차광한 차나무 참녹과 보향 잎의 테아닌 함량은 각각 549.31 ± 48.84 mg/kg과 412.17 ± 47.56mg/kg으로 확인되었으며, 암조건에서 차나무 참녹과 보향 잎의 테아닌 함량은 각각 362.66 ± 12.84 mg/kg과 238.18 ± 28.82mg/kg으로 분석되었다. 차광하지 않은 조건에서 테아닌 함량은 115–127mg/kg 수준으로 유사했다. 차나무 참녹의 차광에 따른 테아닌의 함량변화는 50% 차광조건에서 473%, 암조건에서 312% 증가했다. 또한 차나무 보향의 차광에 따른 테아닌의 함량은 50% 차광조건에서 322%, 암조건에서 186% 증가했다. 차광으로 인한 테아닌 함량의 증가률은 참녹이 보향보다 높은 것으로 분석되어 참녹이 보향보다 차광에 따른 테아닌 합성이 우수한 것을 확인하였다. 암조건에서 테아닌 함량은 50% 차광조건의 테아닌 함량보다 낮게 나타났다. 차나무 잎의 테아닌은 빛 투과율이 감소할수록 테아닌 함량이 증가한다고 알려져 있다(Song et al. 2013; Yang et al. 2021a). 35%부터 95%까지 빛을 차광 차나무에서 테아닌 함량은 빛 투과율이 낮을수록 테아닌 함량이 증가한다고 보고되었다. 그러나, 빛을 100% 차단했을때 테아닌 함량변화에 관해서는 명확하게 규명되지 않았으며, 100% 빛을 차단한 본 연구의 실험조건과 달라 향후 좀 더 심도 있는 연구가 진행되어야 할 것이다. 차광하지 않은 차나무의 테아닌 합성유전자 발현은 다른 조건에 비해 가장 낮은 발현량을 확인할 수 있으며, 테아닌 함량도 이와 유사하게 나타났다. 50% 차광한 차나무 잎의 테아닌 합성유전자는 차광하지 않은 차나무에 비해 150–160%정도 발현량이 증가하였고, 테아닌 함량은 참녹과 보향에서 470%와 320%로 매우 높게 증가하였다. 또한, 암조건에서 차나무의 테아닌 합성유전자는 차광하지 않은 차나무에 비해 186%와 137%의 가장 높은 발현량을 확인하였으나, 테아닌 함량은 참녹과 보향에서 310%와 185%로 증가되어 50% 차광조건보다 낮은 증가량을 보였다. 차나무 품종간의 테아닌 함량을 비교한 결과 잎과 뿌리에서 ‘Huangjinya’, ‘Anjibaicha’, ‘Yingshuang’ 순으로 높다고 보고하였다(Liu et al. 2017). 따라서 테아닌 함량은 품종과 식물조직에 따라 매우 다른 양상이 확인되며, 한 개체에서도 어린잎, 오래된 잎, 뿌리, 줄기 순으로 높은 테아닌 함량을 확인할 수 있다(Selvendran and Selvendran 1973; Jiang 2013). 이에 참녹과 보향의 다른 테아닌 합성유전자 발현과 테아닌 함량 차이는 품종간 특성에 기인한 것이라 사료된다. 또한, 테아닌 합성 경로는 합성, 수송 및 가수분해를 포함한 테아닌 대사 경로의 발견 이후 오랜 시간 연구되었으며, 현재까지 수많은 효소가 테아닌의 대사 경로에 관여하고 있음이 보고되었다. 또한, 차나무에서 테아닌 대사 관련 유전자 발현은 테아닌 함량, 분포 및 대사 경로에 큰 영향을 미칠 수 있음을 확인했다(Deng et al. 2010; Shi et al. 2011). 유전자의 발현은 여러 수준에서 조절되는 복잡한 과정으로 mRNA expression 및 transcription 과정과 protein translation을 통해 단백질의 번역 후 다른 단백질과의 상호작용을 통해 최종 생성물이 생성되는 복잡한 다단계 과정을 거친다(Buccitelli and Selbach 2020). 대사 과정은 다양한 유전자의 발현과 억제를 통한 상호작용이 이루어지고 있다(Schwartz and Ciechanover 2009; Montgomery and Dermitzakis 2011). 또한, mRNA와 단백질 합성과의 관계는 일반적 선형관계로 표현되지만, mRNA 발현이 단백질 합성과정을 촉진하지만, 단백질 합성과정에서 다양한 변수와 복잡성으로 인한 가변성에 의해 단백질 합성의 차이를 보일 수 있다(Buccitelli and Selbach 2020). 이러한 이유로 인해 암조건에서 테아닌 합성유전자의 발현은 가장 높았으나, 테아닌 함량은 50% 차광조건보다 약간 감소했을 것으로 사료된다.
본 연구는 차나무 육종시 유목 단계에서 고품질의 테아닌 함량이 높은 개체를 대량신속검색기술(High Throughput Screening system, HTS system)에 활용가능한 테아닌 선별 분자마커를 개발하고자 하였다. HTS기술은 단시간에 많은 시료를 분석할 수 있는 기술로 유용한 품종을 빠르게 선발할 수 있다. 따라서, 이 기술에 사용되는 probe(molecular marker)의 개발과 검증이 매우 중요하다. 차나무 차광실험을 통해 theanine synthetic gene mRNA의 발현과 테아닌함량을 확인하였다. 빛의 차광으로 인해 테아닌 합성유전자 발현량이 증가하면, 테아닌 함량도 증가되는 것을 확인하였으며, 이 결과를 통해 theanine synthetic gene mRNA의 발현과 테아닌 함량은 매우 높은 상관관계를 갖는 것을 확인하였다. 본 연구에서 설계된 테아닌 합성유전자를 검출하는 probe를 활용해 테아닌 생합성유전자의 발현량을 확인하고, 테아닌 함량이 높은 고품질의 차나무를 조기선별하는 분자마커로 활용할 수 있을 것으로 판단된다.


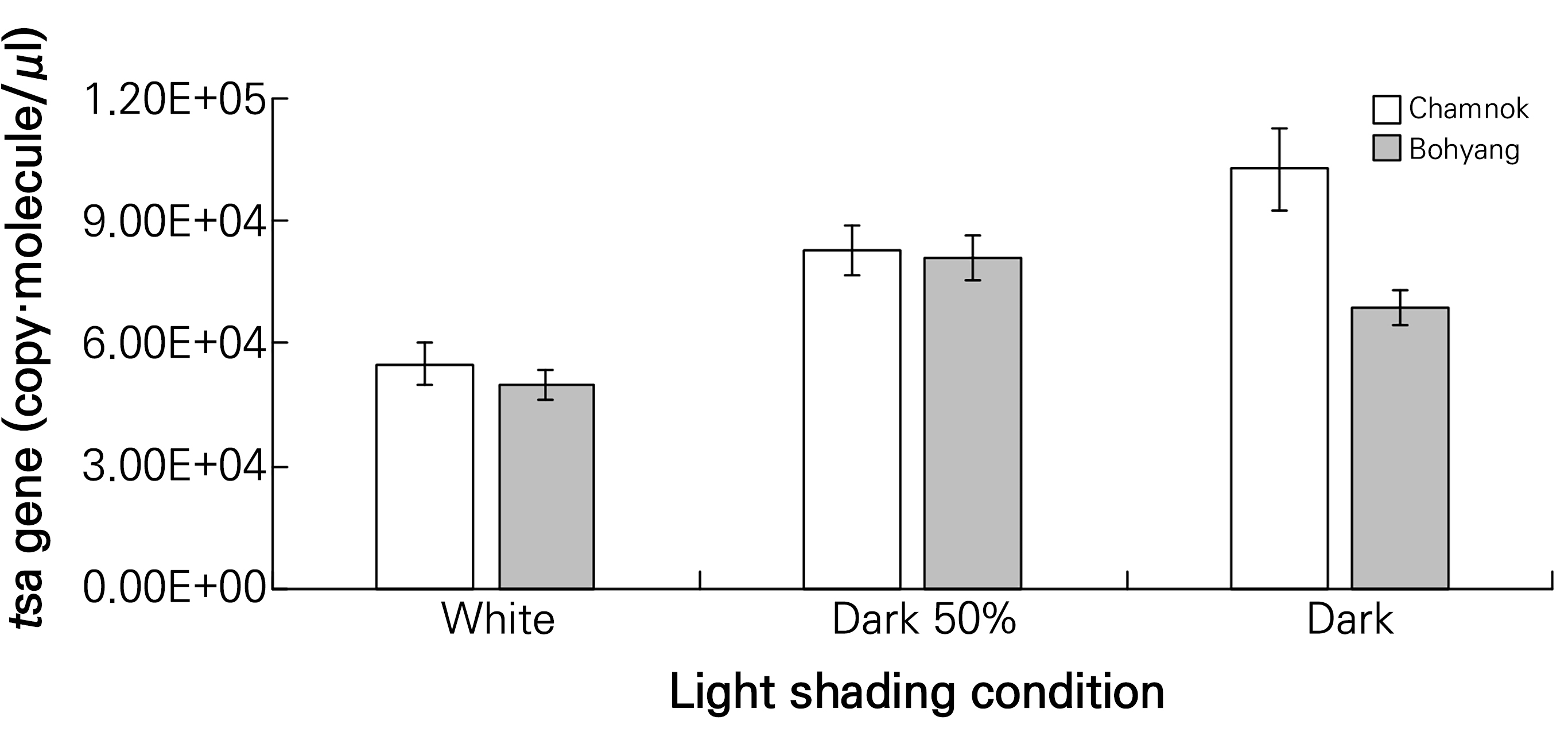
 ; Chamnok,
; Chamnok,  ; Bohyang).
; Bohyang).