서 언
재료 및 방법
춘란 시료의 수집
DNA 추출
Multiplex PCR을 이용한 12 SSR 좌위의 유전자형 결정
SSR 마커에 대한 집단유전학적 특성 분석
유사 복합유전자형의 검색
형매 확률 예측 및 계통학적 분석
결 과
자매품종 그룹에서 관찰된 복합유전자형의 종류 및 빈도
자매품종 그룹내 복합유전자형의 분포 및 빈도
동일 자매품종 그룹에서 관찰된 복합유전자형 상호간의 높은 형매 확률
자매품종 그룹에서 관찰된 복합유전자형들의 계통학적 유연 관계
고 찰
서 언
난초과(Orchidaceae)에 속하는 심비디움(Cymbidium)속은 약 60 종이 중국, 한국, 일본, 인도 및 호주 등 주로 열대 및 아열대 아시아 지역에 분포되어 있다(Dressler, 1993; DuPuy and Cribb, 2007; Motomura et al., 2008; Leitch et al., 2009). Cymbidium 속의 대표적인 종인 춘란(C. goeringii)은 야생에서도 서식하지만, 한국을 포함한 동북아시아에서는 산업적으로 매우 중요하게 재배되는 종 중 하나로, 잎과 꽃의 형태 및 색상의 다양한 변이성을 보인다. 한국에서는 야생에서 채집되어 재배되는 5,000 품종 이상이 대한민국난등록협회(http://www.koreso.com/) 또는 한국난연합회 난등록위원회(http://www.kour.or)에 등록되어 있다.
Cymbidium 속 식물들의 유전적 다양성 및 품종 식별을 위한 여러 종류의 유전적 마커들이 개발되어 왔다. 그 종류들로는 randomly amplified polymorphic DNA (RAPD) (Obara-Okeyo and Kako, 1998; Choi et al., 2006), amplified fragment length polymorphism (AFLP) (Jian and Zhu, 2013), inter-simple sequence repeat (ISSR) (Wang et al., 2009), 및 simple sequence repeat (SSR) (Moe et al., 2010; Huang et al., 2011; Hyun et al., 2012; Lee et al., 2012; Tang et al., 2012; Li et al., 2014; Lee et al., 2020) 등이 보고되어 왔다. 그 중에서도 microsatellite나 short tandem repeat (STR)로도 불리는 초다형성 SSR 좌위는 다중 대립유전자 마커로서, 개체간 유전적 변별력과 실험 조건에 따른 재현성이 우수할 뿐 아니라, 여러 좌위의 동시 PCR 증폭 및 자동염기서열분석기를 이용한 분석이 용이하여, 최근에는 가장 많이 활용된다.
현재 상업적 가치를 가지면서 특정 품종으로 확립된 C. goeringii 변이체의 대부분은 처음에 산에서 특징적인 표현형을 가진 야생 춘란이 채취되어 고정된 후, 여러 난 재배자들에게 퍼진 것이다. C. goeringii의 주요 품종들은 원예학적으로 크게 특징적인 꽃 색상과 모양을 보여주는 “화예품(flower-variant type)”과 특징적인 잎 색상과 무늬 패턴을 보여주는 “엽예품(leaf-variant type)”의 두 유형으로 구분된다. 야생에서 춘란은 주로 유성생식으로 번식하는데, 타가수분과 자가수분이 모두 일어나는 것으로 여겨진다(Lee et al., 2004; Suetsugu, 2015). 그렇지만 야생에서 채취된 후 특정 품종으로 고정된 춘란 품종은 대부분 무성생식에 의해 증식되어 전파된다. 이론적으로, 야생에서 채취된 후 품종으로의 고정 및 전파 과정 동안 발생할 수 있는 돌연변이를 무시할 때, 특정 품종 내의 모든 개체들은 유전적으로 완전히 동일해야 한다.
춘란 시장에서 특정한 품종으로 거래되는 춘란의 상당한 비율이 처음 명명된 품종으로부터 영양번식된 개체가 아닐 수 있는 것으로 보고된 바 있다. Lee 등은 수십 품종의 한국 춘란에 대한 8개의 SSR 마커의 복합유전자형(combined genotypes)을 확립하여 특정 품종을 구별하기 위한 분자유전학적 식별 방법으로서 가능성을 제시하였다(Lee et al., 2012). 최근에는 Lee 등이 춘란의 주요 10 종류의 화예품종을 대상으로 13 좌위에 대한 SSR 복합유전자형을 조사하였는데, 약 40%가 최초 등록된 품종과 유전적으로 다른 것으로 추정되었다(Lee et al., 2020). 춘란 거래의 대부분은 꽃이 피어 있지 않은 상태에서 이루어지기 때문에, 화예품을 잎 모양 등으로만 정확한 품종을 식별하는 일은 전문가들도 쉽지 않은 것으로 알려져 있다.
춘란을 재배하거나 상업적 거래를 하는 사람들은 인접한 야생의 장소에서 채취되어 서로 간에 유사한 표현형적 특징을 보이는 품종들을 일반적으로 서로 “자매종(sister species)”으로 부른다. 진화생물적으로 자매종은 두 종이 진화 과정 동안 분리되었지만, 가장 최근의 공동 조상을 서로 공유하는 계통유전적으로 매우 가까운 종을 의미한다(Padian et al., 1994; Sun et al., 2015). 동일한 지역에 서식하면서 분류학적으로 연관성이 높은 종들에 대해 동소종(sympatric species)이라는 용어를 사용하기도 하는데, 난초과에서 동소종에 대한 연구는 생식, 교잡 및 Mycorrhiza와 같은 균류의 공생 연구에서 상당히 수행되었다(Gervasi et al., 2017; Chen et al., 2019; Sujii et al., 2019). 춘란 품종 전체는 생물학적으로 C. goeringii의 단일 종(species)으로 분류되기 때문에, 춘란 재배자와 상거래 관계자들이 흔히 칭하는 자매종을 본 연구에서는 “자매품종(sister cultivars)”으로 표현하였다.
우리나라에서 서로 자매품종으로 불리는 춘란 품종들은 제법 존재하지만, 해당 품종들의 원 채집지나 채집자에 대한 정확한 정보가 없는 경우가 많아 실제로 자매품종들이 서로 유전적으로 얼마나 가까운 지를 예측하기는 쉽지 않다. 대표적인 자매품종 그룹으로는 ‘홍노을(Hongnoeul)’, ‘루비(Ruby)’, ‘노을(Noeul)’, ‘세홍소(Sehongso)’, ‘구담(Goodam, 태양(Taeyang)으로도 불림)’으로 명명된 품종 그룹으로 전라도 지역에서 처음 채취된 것으로 추정된다. 꽃의 특징은 ‘홍노을’과 ‘노을’은 홍화소심(잎술꽃잎에 반점이 없거나 옅은 붉은 색 계열의 꽃)이지만 ‘루비’, ‘세홍소’, ‘구담’은 주금소심(잎술꽃잎에 반점이 없거나 옅은 주금 색 계열의 꽃)으로 약간의 차이가 있다. 서로 자매품종으로 알려진 ‘정담(Jeongdam, 천상화(Cheonsanghwa)로도 불림)’과 ‘미정소(Mijeongso)’는 경남에서 채취된 것으로 알려져 있으며 두 품종 모두 주금소심으로 비슷한 특징을 보인다. 동일한 자매품종 그룹내 품종들이라도 품종에 따라서는 판매 가격에 차이가 나기도 한다. 아직까지 춘란의 자매품종의 그룹내에서 각 품종들이 서로 유전적으로 얼마나 가까운 지에 대한 근친의 정도를 조사한 연구는 전혀 수행된 적이 없다.
본 연구의 목적은 C. goeringii의 SSR 유전자형이 자매 품종 그룹 내 개별 품종들 사이에서 얼마나 일치하는지를 결정하고, 상호 유전적 연관성을 분석하는 것이다. 본 연구를 위해 4개의 자매품종(혹은 유사한 SSR 복합유전형을 보이는 품종) 그룹으로 구성된 13개 한국 춘란 품종과 외부그룹(outgroup)으로 일본과 중국의 각 1 품종을 수집하고, 12 SSR 좌위의 복합유전자형을 결정하였다. 또한 본 연구는 결합유전자형의 빈도와 분포를 조사하여 각 자매품종 그룹내 품종들 간의 유전적 연관성과 식별력에 대해 분석하였다.
재료 및 방법
춘란 시료의 수집
본 연구는 춘란 재배자 및 상거래자들을 통해 2016년 1월부터 2020년 4월까지 난원 등에서 재배되는 화예품 계통으로 알려진 한국 춘란(C. goeringii) 81 품종으로부터 총 1,850 개체의 시료를 확보하였다. 이 시료들 중에서 서로 자매품종으로 알려져 있거나, SSR 유전자형의 비교 분석을 통해 유전적 근친 가능성이 예측된 춘란품종들에 대해 서로 자매품종 그룹으로 분류하였다. 그 결과 4개 자매품종 그룹에서 13개 품종의 358 시료가 조사 대상으로 선발되었다. 각 자매품종 그룹내 품종들의 꽃의 표현형적 특징, 대한민국난등록협회 등록번호, 최초 채집지에 대한 정보는 Table 1에 제시하였다. 홍화소심이나 주금소심의 표현형을 보이는 자매품종 그룹-1은 5개 품종 124 시료로, ‘홍노을’(45 시료), ‘루비’(14 시료), ‘세홍소’(25 시료), ‘노을’(19 시료), ‘구담’(21 시료)으로 구성되었다(Fig. 1A). 주금소심의 특징을 보이는 자매품종 그룹-2는 3개 품종 43 시료로, ‘정담’(39 시료), ‘미정소’(3 시료), ‘무명’(Anonym, 1 시료)으로 구성되었다(Fig. 1B). 자매품종 그룹-2의 ‘무명’은 최근에 자매품종 그룹-2의 다른 품종이 채취된 장소의 인근에서 채집되어 아직 품종명이 결정되지 않은 주금소심의 화예품종이다. 주금소심의 특징을 보이는 자매품종 그룹-3은 3개 품종 179 시료로, ‘동광’(Donggwang, 95 시료), ‘채운’(Chaeun, 36 시료), ‘홍귀비’(Honggwibi, 48 시료)로 구성되었다(Fig. 1C). 자매품종 그룹-3의 ‘동광’의 95 시료 중 84 시료는 선행 연구에서 보고된 바 있으며(Lee et al., 2020), 11 시료는 본 연구에서 추가하였다. 황화(노란 색 계열의 꽃)의 특징을 보이는 자매품종 그룹-4는 2개 품종 12 시료로, ‘수황’(Soohwang, 10 시료)과 ‘살구’(Salgoo, 2 시료)로 구성되었다(Fig. 1D). 또한 본 연구에서는 외부그룹으로, 일본 품종으로 알려진 황화소심(잎술꽃잎에 반점이 없거나 옅은 노란 색 계열의 꽃)의 ‘호박전’(Hobakjeon, 6 시료)과 중국 품종(C. forrestii)으로 알려진 기화(奇花, 일반적이지 않는 특이한 형태의 꽃)의 ‘녹운’(Nokoon, 11 시료) 시료를 확보하여 SSR 유전자형을 분석하고 비교하였다(Fig. 1E).
Table 1.
Grouping, characterization, and sampling of 4 sister cultivar groups in C. goeringii
| Group | Characteristics of flower phenotypesz |
Number of samples |
Registration Noy |
Original collection placex |
| Cultivar group 1 | ||||
| Hongnoeul | Red flower with non-anthocyanin white lip | 45 | Unregistered | Jeollanamdo |
| Ruby | Orange-yellow flower with non-anthocyanin white lip | 14 | 858 | Unrecorded |
| Sehongso | Orange-yellow flower with non-anthocyanin white lip | 25 | 1191 | Jeollanamdo |
| Noeul | Red flower with non-anthocyanin white lip | 19 | 706 | Unrecorded |
|
Goodam (Taeyang) | Orange-yellow flower with non-anthocyanin white lip | 21 | 19-007 |
Hampyung, Jeollanamdo |
| Subtotal | 124 | |||
| Cultivar group 2 | ||||
|
Jeongdam (Cheonsanghwa) | Orange-yellow flower with non-anthocyanin white lip | 39 | 1617 |
Sacheon, Gyeongsangnamdo |
| Mijeongso | Orange-yellow flower with non-anthocyanin white lip | 3 | 1171 |
Hadong, Gyeongsangnamdo |
| Anonymw | Orange-yellow flower with non-anthocyanin white lip | 1 | Unregistered | Gyeongsangnamdo |
| Subtotal | 43 | |||
| Cultivar group 3 | ||||
| Donggwang | Orange-yellow flower with non-anthocyanin white lip | 95 | 1 | Jeollanamdo |
| Chaeun | Orange-yellow flower with non-anthocyanin white lip | 36 | 150 |
Jangseong, Jeollanamdo |
| Honggwibi | Orange-yellow flower with non-anthocyanin white lip | 48 | Unregistered | Jeollanamdo |
| Subtotal | 179 | |||
| Cultivar group 4 | ||||
| Soohwang | Yellow flower | 10 | 1483 |
Shinan, Jeollanamdo |
| Salgoo | Yellow flower | 2 | 988 |
Buan, Jeollabukdo |
| Subtotal | 12 | |||
| Outgroup | ||||
| Hobakjeon | Yellow flower with non-anthocyanin lip | 6 | Unregistered | Japan |
| Nokoonv (C. forrestii) | Weird flower | 11 | Unregistered | China |
| Subtotal | 17 | |||
| Total | - | 375 | - | - |
zInformation for flower phenotypes were obtained from the Korea Orchid Registration Association (in registered cases).
yRegistration numbers were followed by the Korea Orchid Registration Association, except for ‘Goodam’ registered in the Registration Committee of Korea Orchid Union (http://www.kour.or.kr/).
xCollection places of the corresponding cultivars were followed by the Korea Orchid Registration Association.

Fig. 1.
Flower images of 15 cultivars from four sister cultivar groups and outgroup in C. goeringii. The images were provided from the Korea Orchid Registration Association with permission. A, Sister cultivar group-1; B, Sister cultivar group-2; C, Sister cultivar group-3; D, Sister cultivar group-4; E, Outgroup cultivars.
DNA 추출
DNA 추출을 위한 춘란 시료는 대략 0.5–1.0cm 길이의 각 개체의 잎이나 뿌리를 대부분 사용하였지만 간혹 꽃이나 꽃대를 절취하여 사용하기도 하였다. 20–50mg의 식물 조직을 약 1mm 길이로 짧게 자른 후 막자 사발에 넣고 액체 질소를 공급하면서 미세한 분말로 파쇄하거나, 시료와 용해 완충용액을 넣고 TissueLyser II(Qiagen, Hilden, Germany)를 사용하여 5–7분간 30Hz로 미세 분말로 분쇄하였다. 이어 분쇄된 시료로부터 genomic DNA는 DNeasy Plant Mini Kit(Qiagen)를 사용하여 추출하였으며, DNA 농도는 NanoDrop 2000 Spectrophotometer(Thermo Fisher Scientific, Wilmington, DE, USA)를 사용하여 측정하였다.
Multiplex PCR을 이용한 12 SSR 좌위의 유전자형 결정
본 연구에서는 12 SSR 좌위를 대상으로 복합유전자형을 Lee et al.(2020)의 방법을 약간 변형하여 2 종류의 multiplex PCR 시스템을 사용하여 결정하였다. Primer는 Lee et al.(2020)과 동일한 염기 서열을 사용하였으며, forward primer는 FAM, VIC, NED, PET의 4 종류 형광 염색 물질 중 한 가지로 표지시켜 제작하였다(Bioneer, Daejeon, Korea). 한국 춘란 집단을 대상으로부터 얻은 각 SSR 좌위의 반복 단위, 관찰된 대립유전자 종류, PCR 증폭 DNA의 크기 및 GenBank(http://www. ncbi.nlm.nih.gov/genbank/) 등록번호와 집단유전학적 통계 분석 값은 Table 2와 같다. 첫번째의 Septaplex PCR 시스템은 CG415, CG709, CG722, CG787, CG1023, CG1210 및 CG1281의 7 SSR 마커를 포함하였으며, 두번째의 Pentaplex PCR 시스템은 CG649, CG1028, CG1085, CG1320 및 CG1400의 5 SSR 마커를 포함하도록 제작하였다. Multiplex PCR의 반응 혼합물은 30–50ng의 genomic DNA와 차등 농도로 혼합된 primer mix를 MultiMAX PCR kit(iNtRON Biotechnology, Sungnam, Korea)와 함께 사용하여 총 10µL 반응액으로 제조하였다. PCR은 SimpliAmp Thermal Cycler(Applied Biosystems, Foster city, CA, USA)를 사용하여 Septaplex PCR은 95°C에서 10 min의 예비변성 후 95°C 20sec, 58°C 1min, 72°C 1min의 반복 조건으로, Pentaplex PCR은 95°C에서 10min의 예비변성 후 95°C 20sec, 58°C 40sec, 72°C 1min의 반복 조건으로 각각 35회 증폭한 후, 마지막으로 72°C에서 30min 추가 반응시켰다. PCR로 증폭된 DNA는 SeqStudio Genetic Analyzer (Applied Biosystems)를 사용하여 모세관 전기영동을 실시하여 분리한 후, GeneMapper 프로그램(NT, Ver. 6.0, Thermo Fisher-Applied Biosystems)을 사용하여 각 SSR 마커의 유전자형을 결정하였다. 복합유전자형은 각 시료별 12 SSR 마커의 유전자형을 동시에 표시한 것으로 정의하였다.
Table 2.
Information of 12 C. goeringii SSR markers in the control group
| Locus | Repeat unit | Observed allele number | Heterozygosity |
Polymorphism information content |
Power of discrimination |
PCR size (bp) |
GenBank accession numberz | |
| Hexp | Hobs | |||||||
| Septaplex system | ||||||||
| CG415 | AG | 10 | 0.742 | 0.449 | 0.709 | 0.869 | 110-140 | HQ842905 |
| CG709 | AG | 16 | 0.805 | 0.565 | 0.782 | 0.926 | 148-194 | HQ842922 |
| CG722 | AG | 13 | 0.763 | 0.537 | 0.728 | 0.899 | 160-200 | HQ842923 |
| CG787 | AG | 11 | 0.664 | 0.269 | 0.636 | 0.765 | 104-128 | HQ842926 |
| CG1023 | AG | 14 | 0.497 | 0.267 | 0.467 | 0.640 | 188-224 | HQ842937 |
| CG1210 | AG | 17 | 0.751 | 0.510 | 0.723 | 0.887 | 104-162 | HQ842946 |
| CG1281 | AG | 9 | 0.591 | 0.322 | 0.545 | 0.731 | 145-189 | HQ842953 |
| Pentaplex system | ||||||||
| CG649 | AG | 21 | 0.763 | 0.445 | 0.748 | 0.880 | 89-151 | HQ842919 |
| CG1028 | AG | 17 | 0.759 | 0.449 | 0.735 | 0.871 | 114-162 | HQ842938 |
| CG1085 | AG | 17 | 0.584 | 0.290 | 0.559 | 0.715 | 80-148 | HQ842942 |
| CG1320 | CAA | 11 | 0.704 | 0.370 | 0.704 | 0.839 | 125-152 | HQ842954 |
| CG1400 | AG | 15 | 0.812 | 0.517 | 0.783 | 0.920 | 195-257 | HQ842958 |
| Mean | - | 14.2 | 0.703 | 0.416 | 0.677 | 0.828 | - | - |
SSR 마커에 대한 집단유전학적 특성 분석
각 SSR 마커에 대한 대립유전자의 명명은 Bär et al.(1997)과 Hyun et al.(2012)의 방법에 따라 SSR 좌위별 반복 서열의 반복 횟수로 결정하였다. SSR 좌위의 집단유전학적 특성을 분석하기 위해서는 복합유전자형이 서로 다른 재배 춘란의 147 시료를 선별하여 활용하였으며, 이형접합도(heterozygosity), 다형성 정보 지수(polymorphic information content: PIC), 변별력(power of discrimination: PD)은 Cervus 프로그램(Ver. 3.0.7, Field Genetics Ltd, London, UK, http://www.fieldgenetics. com/)과 PowerStatsV12 프로그램(Promega, Madison, WI, USA: http://www.promega.com/geneticidtools/)을 사용하여 결정하였다.
유사 복합유전자형의 검색
많은 복합유전자형으로부터 특정 복합유전자형과 유사성을 가지는 것을 효율적으로 비교 검출하기 위해서는 MATLAB (MathWorks, Inc., Natick, MA, USA)으로 간단한 프로그램을 제작하여 사용하였다. 이 프로그램은 복합유전자형이 기록된 데이터베이스를 읽어서 시료의 복합유전자형 간의 거리를 계산하고, 이 거리를 기반으로 시료들의 유사성을 비교하였다. 프로그램상에서 (1) 시료 데이터베이스 상의 품종명을 입력하거나 (2) 찾고 싶은 복합유전자형을 직접 검색하면, 데이터베이스 상에서 복합유전자형 간의 거리가 가까운 시료 순으로 일치하는 검색 결과를 보여준다. 복합유전자형 간의 거리는 Rogers의 유전적 거리 계산 방식을 보다 단순화한 것으로, 총 12개 SSR 마커 각각에 대해서 두 시료의 유전자형이 같으면 0, 다르면 1을 더하여 산출하였다(Rogers, 1972). 즉, 복합유전자형이 완전히 일치하면 거리가 0이 되며, 완전히 불일치할 경우의 거리는 12가 되도록 설정하였다.
형매 확률 예측 및 계통학적 분석
형매 확률(sibling probability)을 결정하기 위해서는 집단유전학적 특성 분석에 사용된 12 SSR 좌위에 대한 서로 다른 복합유전자형을 가진 81개 품종(147 시료)을 활용하였으며, SSR 좌위 별 유전자형의 빈도 값으로 Bayes 이론을 적용하여 각각의 복합유전자형들 간의 형매 확률을 결정하였다(Evett and Weir, 1998). SSR 복합유전자형에 대한 유전적 거리는 GenAlEx v6.5 통계 프로그램을 사용하여 결정하였다(Peakall and Smouse, 2012). 비유사성의 매트릭스(matrix of dissimilarities)로부터 비가중 산술 평균 결합법(unweighted pair-group method with arithmetic average: UPGMA)을 사용하여 계통도(phylogenetic dendrogram)를 작성하였다. 주성분 분석(principal component analysis)에서 거리 매트릭스는 각각의 복합유전자형을 가진 C. goeringii 개체 간의 유전적 연관성을 그래프로 표현하는데 사용되었다.
결 과
자매품종 그룹에서 관찰된 복합유전자형의 종류 및 빈도
본 연구에서 분석된 춘란 대조 집단의 147 시료에 대한 12 SSR 좌위의 평균 대립유전자 수는 14.2개였으며, 예측이형접합도(Hexp), PIC 및 PD의 평균 값은 각각 0.703, 0.677, 0.828로 계산되었다(Table 2). 특히 임의의 두 개체 간에 12 마커 모두의 유전자형이 우연히 동일할 확률을 의미하는 일치율(matching probability)은 1.4 × 10-10으로 매우 높았다. 이와 같은 높은 다형성 및 변별력은 본 연구에서 사용된 SSR 좌위들이 개체간 혹은 품종 간의 유전적 구분을 위해 유용하게 활용될 수 있음을 보여주었다. 본 연구에서 사용된 SSR 복합유전자형은 춘란의 품종들을 유전적으로 구분할 수 있는 가능성이 선행 연구에서 제시된 바 있다(Lee et al., 2012, 2020). 그런데, 관측이형접합도(Hobs)의 평균값이 0.416으로 Hexp의 평균값(0.703)과 비교하면 상당한 수준으로 떨어지는 것으로 조사되었다(Table 2). 이런 현상은 PCR primer 부착 서열에서의 변이성에 기인한 일부 대립유전자의 증폭 실패(allele dropout)로 실제 이형접합자가 동형접합자로 관찰되는 현상이 가장 큰 원인으로 예상되지만(Hahn et al., 1998), 사용한 춘란 시료들의 근친교배 가능성도 한 원인으로 예상된다.
본 연구는 여러 춘란 품종들로 구성된 시료들의 복합유전자형을 비교 분석하여, 총 13개 품종의 358 개체를 포함하는 4개 자매품종 그룹에서 13 종류의 복합유전자형을 분리하였다(Table 3). SSR 12 좌위에 대한 복합유전자형은 각 좌위별 대립유전자 쌍으로 표기하였으며, 각 그룹별 복합유전자형의 분포는 Fig. 2A에 나타내었다.
Table 3.
Combined genotypes of 12 SSRs observed in four cultivar groups and outgroup
|
Cultivar group |
Combined genotype | Genotype of each SSR markerz | |||||||||||
| CG415 | CG649 | CG709 | CG722 | CG787 | CG1023 | CG1028 | CG1085 | CG1210 | CG1281 | CG1320 | CG1400 | ||
| Group-1 | CG1-1 | 13-13 | 15-15 | 17-18 | 11-22 | 24-24 | 28-28 | 7-7 | 14-14 | 6-6 | 9-9 | 14-14 | 20-23.1 |
| CG1-2 | 13-13 | 29.1-29.1 | 17-18 | 11-22 | 24-24 | 28-28 | 17-17 | 14-14 | 29-29 | 9-10 | 10-10 | 23.1-23.1 | |
| CG1-3 | 13-13 | 15-15 | 17-18 | 11-11 | 24-24 | 28-28 | 7-17 | 14-18 | 29-29 | 9-10 | 10-10 | 20-23.1 | |
| CG1-4 | 13-13 | 15-29.1 | 18-18 | 11-22 | 24-24 | 28-28 | 17-17 | 18-18 | 29-29 | 9-10 | 10-14 | 20-23.1 | |
| CG1-5 | 13-13 | 15-15 | 17-17 | 11-17 | 24-24 | 20-20 | 17-18 | 14-14 | 15-17 | 9-16 | 7-12 | 20-20 | |
| Group-2 | CG2-1 | 13-15 | 15-29.1 | 18-25 | 21-21 | 24-24 | 13-13 | 17-17 | 15-15 | 12-15 | 16-16 | 7-7 | 20-20 |
| CG2-2 | 15-15 | 25.1-29.1 | 18-25 | 12-12 | 24-24 | 13-13 | 17-17 | 15-15 | 12-15 | 16-16 | 7-7 | 19-20 | |
| Group-3 | CG3-1 | 13-13 | 15-15 | 12-16 | 12-12 | 22-24 | 13-20 | 15-17 | 14-14 | 6-17 | 9-16 | 7-13 | 13.1-20 |
| CG3-2 | 13-13 | 15-29.1 | 12-16 | 12-17 | 24-24 | 13-20 | 15-15 | 14-14 | 6-17 | 9-9 | 7-7 | 20-20 | |
| CG3-3 | 13-13 | 29.1-29.1 | 16-16 | 12-12 | 22-24 | 13-13 | 15-15 | 14-14 | 17-17 | 9-16 | 7-13 | 13.1-20 | |
| Group-4 | CG4-1 | 13-13 | 14-21 | 17-20 | 11-12 | 24-24 | 13-13 | 17-17 | 14-14 | 15-16 | 16-16 | 7-12 | 13.1-14 |
| CG4-2 | 13-13 | 15-21 | 17-17 | 11-12 | 24-24 | 13-13 | 15-17 | 12-12 | 15-16 | 16-16 | 12-12 | 14-15 | |
| CG4-3 | 13-13 | 14-15 | 17-20 | 12-20 | 24-24 | 13-13 | 7-15 | 12-22 | 15-16 | 16-16 | 10-12 | 13.1-19 | |
| Outgroup | Hobakjeon | 18-22 | 17-17 | 22-22 | 16-17 | 17-25 | 20-28 | 17-17 | 20-20 | 13-15 | 9-9 | 7-8 | 14-23.1 |
| Nokoon | 11-18 | 17-21 | 20-20 | 10-11 | 12-25 | 6-6 | 6-16 | 11-21 | 9-18 | 10-18 | 8-12 | 14-17 | |
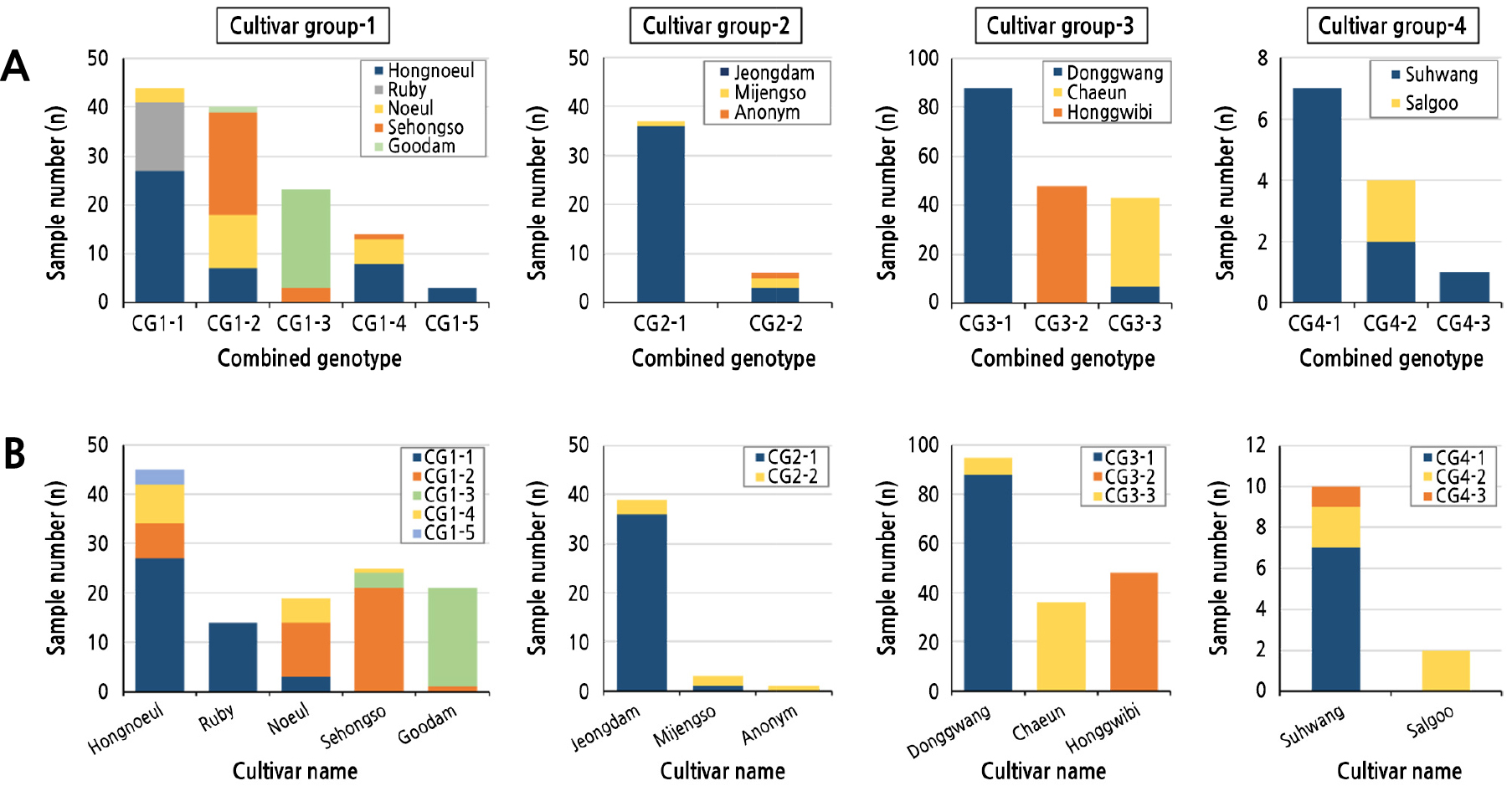
Fig. 2.
Frequencies and distribution of the combined genotypes of 12 SSR markers among cultivar groups in C. goeringii. A, Observed sample number of each cultivar according to combined genotypes in each cultivar group; B, Observed sample number of each combined genotype according to cultivars in each cultivar group.
자매품종 그룹-1
‘홍노을’, ‘루비’, ‘세홍소’, ‘노을’, ‘구담(태양)’으로 구성된 자매품종 그룹-1의 124 시료에서 서로 유사하며 근친성이 추정되는 5개의 복합유전자형이 관찰되었다. 그룹-1의 시료 중, CG1-1 복합유전자형은 44 시료에서 관찰되어 가장 높은 빈도(35.5%)를 보였으며, 두번째로 높은 빈도를 보인 CG1-2 복합유전자형은 40 시료(32.3%)에서 관찰되었다. 이어 CG1-3과 CG1-4 복합유전자형은 각각 23 시료(18.5%)와 14 시료(11.3%)에서 관찰되었고, 가장 낮은 빈도의 CG1-5 복합유전자형은 3 시료(2.4%)에서만 관찰되었다. 각 복합유전자형들은 서로 상당히 유사하여, 그룹-1의 모든 품종들은 유전적으로 가까운 근친일 가능성이 제시하였다.
자매품종 그룹-2
‘정담(천상화)’, ‘미정소’ 및 ‘무명’을 포함하는 자매품종 그룹-2의 43 시료에서는 서로 유사하며 근친성이 추정되는 2개의 복합유전자형만이 관찰되었다. CG2-1 복합유전자형은 37 시료(86.0%)에서 관찰되어 상당히 높은 빈도를 나타냈으며, CG2-2 복합유전자형은 6 시료(14.0%)에서만 관찰되었다.
자매품종 그룹-3
자매품종 그룹-1 및 -2와 다르게, 그룹-3의 ‘동광’, ‘채운’ 및 ‘홍귀비’가 인접한 지역에서 채취되어 각각의 품종으로 고정되었다는 직접적인 근거는 없는 실정이다. 그렇지만, 본 연구에서는 3 품종 모두 전라남도에서 채취된 것으로 알려져 있으며, 품종 간 복합유전자형의 유사성이 높아 서로 근친으로 의심되어 자매품종으로 묶어서 유전자형 분석을 수행하였다. 그룹-3의 총 179 시료에 대한 SSR 좌위 분석에서 서로 유사하며 근친성이 추정되는 3개의 복합유전자형이 관찰되었다. 가장 높은 빈도를 보인 CG3-1 복합유전자형은 약 절반인 88 시료(49.2%)에서 관찰되었으며, CG3-2와 CG-3-3 복합유전자형은 각각 48 시료(26.8%)와 43 시료(24.0%)로 비슷한 빈도로 관찰되었다.
자매품종 그룹-4
자매품종 그룹-4의 ‘수황’과 ‘살구’도 그룹-3과 마찬가지로 비슷한 지역에서 채취되어 품종으로 고정되었다는 직접적인 근거는 없다. 그렇지만, 본 연구에서는 두 품종 모두 전라도 지역에서 채취한 것으로 알려져 있으며, 품종 간 복합유전자형의 유사성이 높아 서로 근친으로 의심되어 자매품종으로 복합유전자형을 분석하였다. 그룹-4의 12 시료에서는 서로 유사하여 근친성이 추정되는 3개의 복합유전자형이 관찰되었다. 가장 높은 빈도의 CG4-1과 두번째 높은 빈도의 CG4-2 결합유전자형은 각각 7 시료(58.3%)와 4 시료(33.3%)에서 관찰되었으며, CG4-3 복합유전자형은 단지 1 시료(8.3%)에서만 관찰되었다.
자매품종 그룹내 복합유전자형의 분포 및 빈도
각 자매품종 그룹내 복합유전자형의 품종 간 분포 및 빈도는 Fig. 2B에 나타내었다. 자매품종 그룹 내에서 관찰된 각각의 복합유전자형은 특정 품종에서만 관찰되거나 혹은 매우 높은 빈도로 관찰되는 품종-특이성을 보이는 경우가 많았다. 예를 들어 그룹-1의 CG1-1은 ‘루비’와 ‘홍노을’에서 가장 높은 빈도로 관찰되었지만, CG1-2는 ‘세홍소’와 ‘노을’에서 높았으며, CG1-3은 가장 높은 빈도를 나타낸 ‘구담’에서 대부분 관찰되었다. 그룹-2의 CG2-1은 ‘정담’에서 높은 빈도로 관찰되는 반면, CG2-2는 ‘미정소’에서 가장 높게 관찰되었다.
자매품종 그룹-1
자매품종 그룹-1에서 5개의 복합유전자형이 관찰되었다. CG1-1은 본 그룹에서 35.5%로 가장 높은 빈도를 보여준 복합유전자형으로, ‘홍노을’의 60.0%, ‘루비’의 100%를 차지하였지만 ‘세홍소’와 ‘구담’에서는 전혀 관찰되지 않았다. ‘루비’의 경우 특이하게 14 시료 모두 CG1-1의 단일 복합유전자형만을 보였다. CG1-2는 32.3%의 빈도로 두번째 높은 빈도로 관찰된 복합유전자형으로, ‘세홍소’는 84.0%로 대부분이 CG1-2를 보였으며, ‘노을’에서도 57.9%로 비교적 높은 빈도를 보였다. 그렇지만, ‘홍노을’과 ‘구담’에서는 각각 15.6%와 4.8%의 낮은 빈도를 나타내었고, ‘루비’는 한 시료에서도 관찰되지 않았다. CG1-3의 그룹내 평균 빈도는 18.5%로 ‘구담’의 대부분(95.2%)과 ‘세홍소’의 일부(12.0%)에서만 관찰되었으며, ‘홍노을’, ‘루비’, ‘노을’의 시료에서는 전혀 관찰되지 않았다. CG1-4는 그룹-1에서 11.3%의 비교적 낮은 빈도로 관찰된 복합유전자형으로, ‘홍노을’(17.8%), ‘노을’(26.3%), ‘세홍소’(4.0%)에서만 관찰되어, 어느 품종에서도 가장 높은 빈도를 보이지 않았다. 그룹-1에서 2.5%의 가장 낮은 빈도를 보인 CG1-5는 오로지 ‘홍노을’(6.7%)에서만 관찰되었다. 각 자매품종 별 복합유전자형의 분포 순서는 다음과 같다. ‘홍노을’은 CG1-1 (60.0%) > CG1-4 (17.8%) > CG1-2 (15.6%) > CG1-5 (6.7%) > CG1-3 (0.0%), ‘루비’는 CG1-1 (100%) > CG1-2, CG1-3, CG1-4 및 CG1-5 (0.0%), ‘노을’은 CG1-2 (57.9%) > CG1-4 (26.3%) > CG1-1 (15.8%) > CG1-3 및 CG1-5 (0.0%), ‘세홍소’는 CG1-2 (84.0%) > CG1-3 (12.0%) > CG1-4 (4.0%) > CG1-1 및 CG1-5 (0.0%), ‘구담’은 CG1-3 (95.2%) > CG1-2 (4.8%) > CG1-1, CG1-4 및 CG1-5 (0.0%)로 관찰되었다.
자매품종 그룹-2
2개의 복합유전자형만 관찰된 자매품종 그룹-2에서 CG2-1은 ‘정담’에서 매우 높게 관찰되었으며(92.3%), ‘미정소’에서는 33.3%로 낮게 관찰되었다. 반면, CG2-2는 ‘정담’에서 7.7%로 낮게 관찰되고, ‘미정소’에서 66.7%로 높게 관찰되었다. 그룹-2의 ‘무명’ 1 시료의 복합유전자형은 CG2-2와 완전히 동일하였다. 각 품종 별 복합유전자형의 분포도를 요약하면, ‘정담’은 CG2-1 (92.3%) > CG2-2 (7.7%), ‘미정소’는 CG2-2 (66.7%) > CG2-1 (33.3%), ‘무명’은 CG2-2 (100.0%)로 관찰되었다.
자매품종 그룹-3
자매품종 그룹-3에서는 3 종류의 복합유전자형이 관찰되었는데, 품종 간에 뚜렷한 복합유전자형 분포도의 차이를 보여주었다. CG3-1의 복합유전자형은 ‘동광’에서 92.6%로 매우 높게 관찰된 반면, ‘채운’과 ‘홍귀비’에서는 전혀 관찰되지 않았다. CG3-2의 복합유전자형은 ‘홍귀비’의 전체 시료(100%)에서 관찰되고, 다른 품종에서는 전혀 관찰되지 않았다. CG3-3은 ‘채운’ 전체 시료(100%)와 ‘동광’의 일부 시료(7.4%)에서 관찰된 반면, ‘홍귀비’에서는 관찰되지 않았다. 그룹 3의 품종 별 복합유전자형의 분포도는 다음과 같았다. ‘동광’은 CG3-1 (92.6%) > CG3-3 (7.4%) > CG3-2 (0.0%), ‘채운’은 CG3-3 (100.0%) > CG3-1 및 CG3-2 (0.0%), ‘홍귀비’는 CG3-2 (100.0%) > CG3-1 및 CG3-3 (0.0%)으로 관찰되었다.
자매품종 그룹-4
3 종류의 복합유전자형이 관찰된 자매품종 그룹-4에서 CG4-1 복합유전자형은 ‘수황’의 70%에서 관찰되고, ‘살구’에서는 관찰되지 않았다. CG4-2 복합유전자형은 ‘수황’의 20%와 ‘살구’의 전체 시료(100%)에서 관찰되었으며, CG4-3은 ‘수황’의 한 시료(10.0%)에서만 관찰되었다.
동일 자매품종 그룹에서 관찰된 복합유전자형 상호간의 높은 형매 확률
SSR 좌위의 각 유전자형 빈도값을 기반으로 13 종류의 복합유전자형들 상호간의 형매 확률을 결정하였는데, 동일 자매 그룹에서 관찰된 복합유전자형들은 서로간에 대부분 99.99% 이상의 높은 확률을 보였다(Table 4). 자매품종 그룹-1에서 CG1-1부터 CG1-4까지 4가지 복합유전형은 서로 99.99% 이상의 매우 높은 형매 확률을 보였다. 자매품종 그룹-2에서 관찰된 두 복합유전자형은 서로 99.9954%의 확률을 보였으며, 자매품종 그룹-3의 세 복합유전자형은 서로 간에 99.9955–99.9999%의높은 확률을 보였다. 자매품종 그룹-4의 세 복합유전자형들도 서로간에 99.9995–99.9999%의 역시 높은 확률을 보였다. 다만, 그룹-1의 CG1-5 복합유전자형은 동일 그룹의 다른 네 복합유전자형과의 형매 확률이 낮게 나타났다. CG1-1 및 CG1-3과의 형매 확률은 약 82%로 약간 낮았지만, CG1-2 및 CG1-4와의 형매 확률은 10-17%로 상당히 낮게 나타났다. 그룹-1의 CG1-5 복합유전자형을 제외한 높은 형매 확률은 그룹내 품종들은 서로 동일 부모를 가지거나, 그렇지 않더라도 유전적 근친성이 매우 강함을 의미한다.
Table 4.
Sibling probability (%) between individuals with specific combined genotypes in C. goeringii
서로 다른 그룹의 복합유전자형과 비교하였을 때, 형매 확률은 대부분 10% 이하로 계산되었다. 절반 이상은 1% 미만으로 매우 낮게 나타났지만, 일부 예외적인 경우도 관찰되었다. 그룹-1의 CG1-5는 그룹-1의 다른 복합유전자형들간에는 10–82%의 비교적 낮은 확률을 보였지만, 오히려 그룹-3의 CG3-2와 94.4219%의 상당히 높은 확률을 보였다. 그룹-2의 CG2-2의 경우도 그룹-3의 CG3-3 간에 85.8530%의 비교적 높은 값이 관찰되었다.
자매품종 그룹에서 관찰된 복합유전자형들의 계통학적 유연 관계
자매품종으로 알려져 있거나 근친일 가능성이 있는 복합유전자형들을 대상으로 계통학적 유연 관계를 분석하였다. UPGMA 클러스터는 C. goeringii의 4 그룹에서 유래한 13 종류의 복합유전형과 외부그룹으로서 원산지가 일본인 ‘호박전’ 및 중국인 ‘녹운’의 복합유전자형 각각의 유전적 거리에 대한 매트릭스로부터 작성하였다(Fig. 3). 각각의 유전자형에 대한 dendrogram에서 각 자매품종에서 유래한 복합유전자형들 간에 상관계수는 상당히 높게 나타났다. 각각의 자매품종 그룹에서 유래한 복합유전자형들은 서로 확연히 구분될 수 있는 clustering으로 분리되었는데, 이것은 서로 유전적 거리의 연관성이 매우 높음을 보여준다. 동일 그룹내 다른 복합유전형들 간에 형매 확률이 낮았던 그룹-1의 CG1-5 복합유전자형은 dendrogram에서 비록 그룹-1의 다른 복합유전자형과 클러스터는 이뤘지만, 유전적 거리는 역시 상대적으로 멀게 나타났다. 일본과 중국 기원의 ‘호박전’과 ‘녹운’ 품종에서 관찰된 복합유전자형은 한국 기원의 각 품종에서 유래한 복합유전자형들과는 계통유전학적으로 확연히 분리되었다.
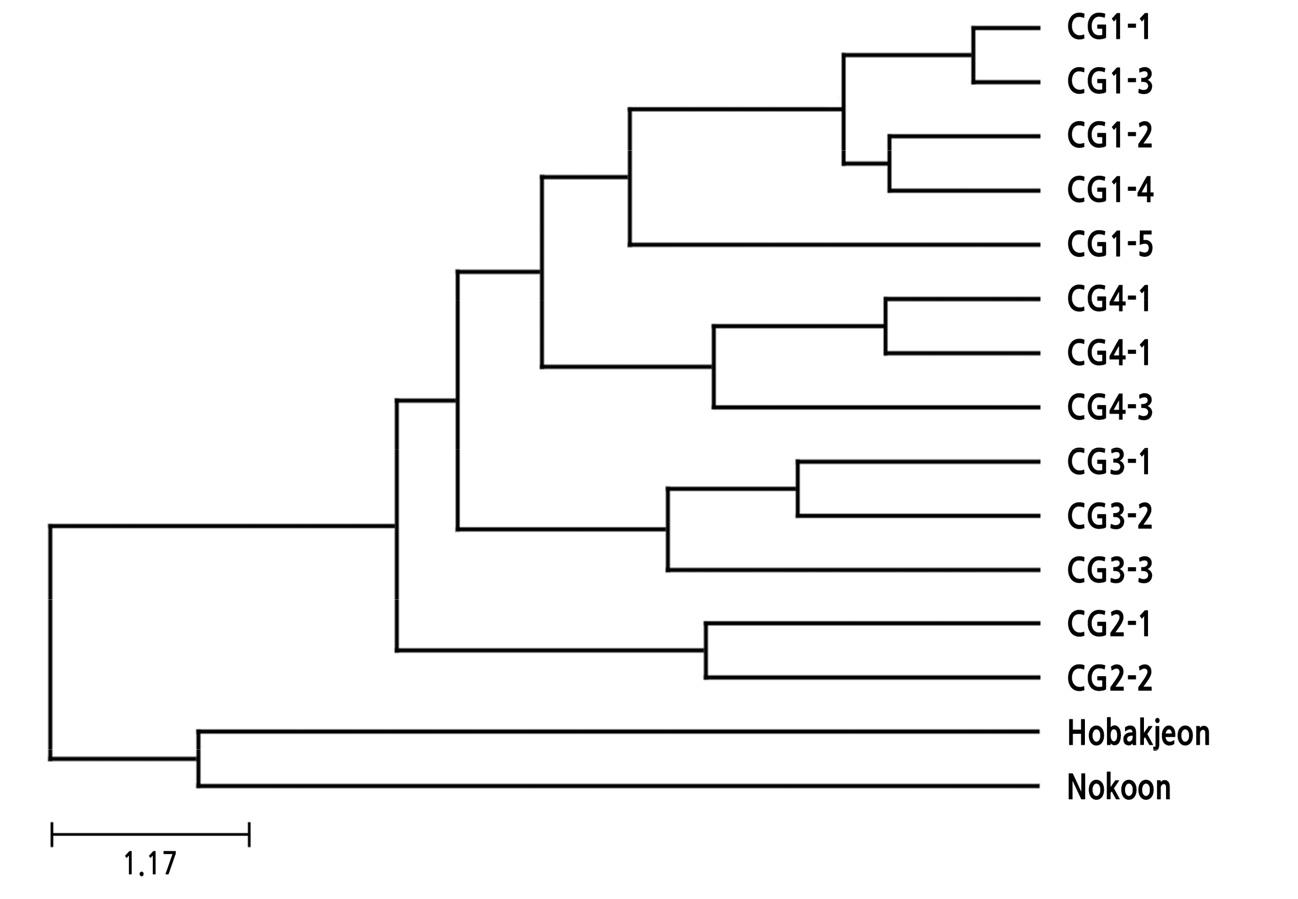
Fig. 3.
Phylogenetic dendrogram of 15 combined genotypes observed from four cultivar groups and outgroup in C. goeringii and C. forrestii. The genetic distance was calculated from the genotypes of 12 SSR loci using the unweighted pair group method with arithmetic average (UPGMA) as the cluster method. The combined genotypes derived from the cultivar group-1, -2, -3 and -4 are indicated by red, green, blue and orange letters, respectively. ‘Hobakjeon’ and ‘Nokoon’ as outgroup are indicated by black letters. The dendrogram shows that the combined genotypes derived from same cultivar groups are clearly clustered into each subgroup.
외부그룹을 포함하여 관찰된 15 종류의 복합유전자형은 주성분 분석(PCoA) 그래프의 두 좌표에 따라서 분류하였을 때, Coord. 1과 Coord. 2의 분별력은 22.78%와 19.21%로 대체로 낮게 나타났다(Fig. 4). PCoA 결과는 Fig. 3의 UPGMA 분석에 의한 계통도 구조와 대체로 일치하였지만, 다소 차이도 관찰되었다. 그룹-1의 5 종류의 복합유전자형은 다른 그룹에 비해 비교적 잘 분리되었지만, 그룹-2, -3, 및 -4에서 유래한 복합유전자형들은 서로 간에 변별력은 낮게 분리되었다. 일본 품종의 ‘호박전’과 중국 품종의 ‘녹운’에서 유래한 복합유전자형은 Coord. 1 및 Coord. 2에 의해 한국 품종들의 복합유전자형들과는 뚜렷하게 분리되었다.
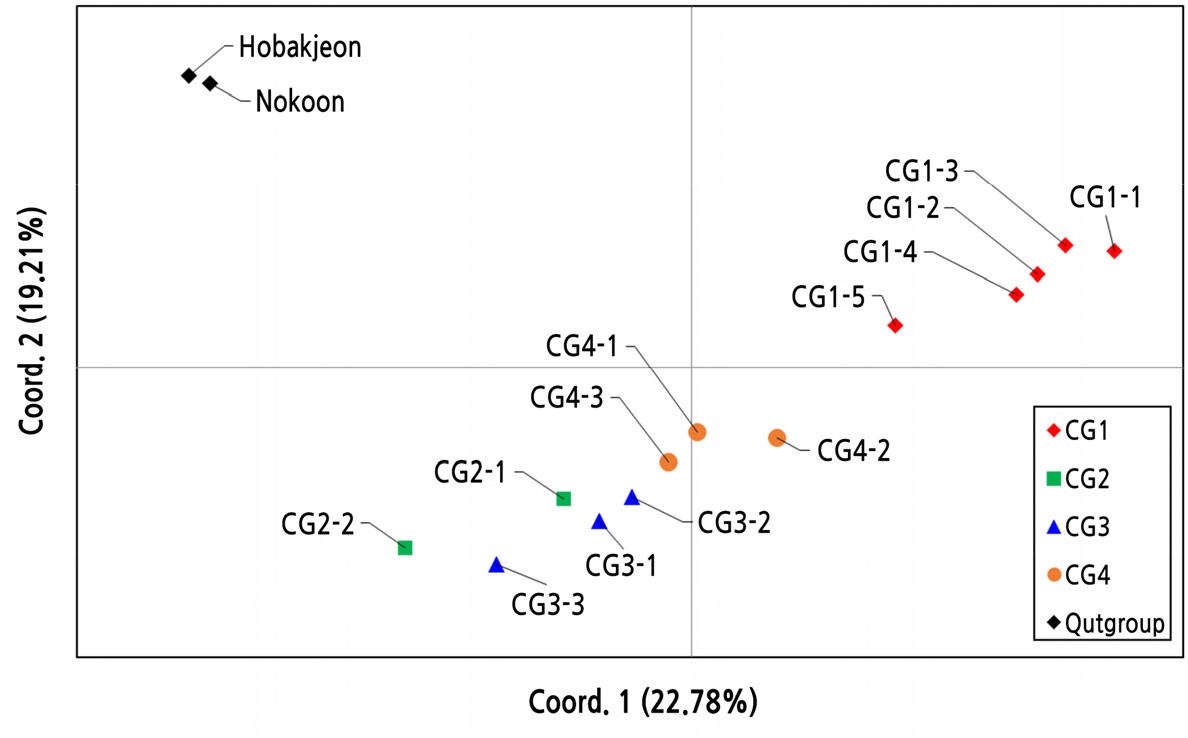
Fig. 4.
Principal coordinate analysis (PCoA) of combined genotypes observed from four sister cultivar groups in C. goeringii. The first two coordinates explained 22.78% and 19.21% of the total variation. The combined genotypes derived from the sister cultivar group-1, -2, -3 and -4 are indicated by red rhombus, green square, blue triangle and orange circle, respectively. Outgroup cultivars of ‘Hobakjeon’ and ‘Nokoon’ are indicated by black rhombus symbol.
고 찰
본 연구는 춘란의 자매품종이나 자매품종으로 추정되는 4 그룹에 대한 12 SSR 좌위의 복합유전자형 조사를 통해 각 그룹내 품종들 간의 근친성(kinship)을 처음으로 분석하였다. 그룹-1에서 5 종류, 그룹-2에서 2 종류, 그룹-3 및 그룹-4에서 3 종류의 복합유전자형이 분리되었다. 각 그룹내 품종에서 관찰된 복합유전자형들은 예상대로 서로 가까운 근친 관계인 것으로 추정되었다. 이와 같은 높은 근친성은 춘란의 높은 자가수분(self-pollination)의 특성과도 연관이 될 것으로 여겨진다.
본 연구에서 분류한 4개 그룹에서 관찰된 13개 복합유전자형에 대한 계통도 분석의 dendrogram은 각 그룹에서 유래된 복합유전자형들 간에 서로 클러스터를 형성하며 유전적으로 상당히 유사함을 보여주었다(Fig. 3). 이런 결과는 각 그룹을 구성하는 개체들이 유전적으로 서로 가까운 근친일 가능성이 있음을 강력하게 시사한다. 각 그룹내 복합유전자형들에 대한 형매 확률을 분석했을 때도 대부분 서로 99.99% 이상의 높은 확률을 보였으며, 다른 그룹 간에 형매 확률은 낮았다(Table 4). 따라서, 유사한 표현형과 복합유전자형을 가지며 서로 인근 지역에서 채집된 자매 품종들은 아마도 부모와 자식 관계(parent-progeny relationship)이거나 형제자매(siblings), 혹은 가까운 근친으로서 특정 꽃의 표현형을 나타내는 유전자형을 공유하는 것으로 예상된다.
각 그룹내 자매품종들의 꽃의 표현형은 서로 비슷하거나 약간의 차이를 보인다. 이런 그룹내 품종 간 표현형적 차이는 꽃의 색소 발현이나 화형에 영향을 주는 다른 2차적인 유전적 요인이나 환경적 요인이 작용한 결과일 수 있다. 최근에 춘란의 개화 기전 및 화형(floral patterning)과 관련된 유전 좌위의 분리 및 전사체(transcriptome) 분석 등의 유전체 연구가 수행되고 있어, 화예품 표현형에 직·간접적으로 영향을 주는 유전자의 분리 및 분자생물학적 기전 규명이 기대되고 있다(Huang et al., 2012; Xiang et al., 2012; Yang et al., 2017; Ramya et al., 2019).
각 그룹내 품종들이 보여주는 복합유전자형의 분포는 자매품종 그룹에 따라 상당한 차이를 보였다. 그룹-1은 특히 자매품종과 복합유전자형이 서로 상당히 혼재된 양상을 보였다. ‘루비’의 모든 시료가 CG1-1의 복합유전자형을 보였으나 ‘홍노을’ 시료의 약 60%도 CG1-1 복합유전자형을 보였으므로, CG1-1을 ‘루비’-특이적, 혹은 ‘홍노을’-특이적 복합유전자형으로 판단하기 어렵다. 비록 난등록협회에 ‘홍노을’과 ‘루비’는 각각 홍화와 주금소심으로 기록되었지만, 두 품종 모두 홍화와 주금화의 중간 정도인 주홍색의 소심으로 비슷한 특징을 보이므로 표현형적 구분이 어렵다. CG1-2 복합유전자형의 경우는 ‘세홍소’의 대부분(84%)과 ‘노을’의 약 58%에서 관찰된 반면, 다른 자매품종에서는 낮게 관찰되었다. ‘세홍소’와 ‘노을’은 각각 주금소심과 홍화소심으로 기록되었지만, ‘노을’이 주금색에 가까운 주홍화를 보이므로, 두 품종 간의 표현형적 구분이 역시 어렵다. 또한, 그룹-1의 품종들은 개체들에 따라 꽃의 표현형이 약간씩의 차이가 있는 것으로 알려져 있다. 이런 차이는 환경적 요인과 더불어, 품종 별 유전적으로 다른 개체들이 혼합되어 있을 가능성을 제시한다. 그룹-1에 대한 복합유전자형과 품종 간의 흐름을 보여주는 alluvial diagram에서도 볼 수 있듯이(Fig. 5), 높은 유전적 근친성과 표현형적 유사성에 기인하여, 각각의 자매품종들로 재배되고 있는 춘란 개체들이 서로 상당히 혼재되었을 가능성을 강하게 제시한다. 그런데, ‘구담’의 경우는 흥미롭게 대부분(95%)이 CG1-3 복합유전자형을 보이므로(CG1-3의 약 87%가 ‘구담’임), CG1-3은 ‘구담’-특이적 복합유전자형으로 여겨진다. 이런 연구의 결과는 그룹-1에서 ‘구담’을 제외한 자매품종들에 대한 세밀한 표현형과 유전자형의 병행 분석을 통한 품종 별 정확한 구분 기준 마련과 품종 분류의 재정립이 필요하다는 것을 제시한다.

Fig. 5.
Alluvial diagram showing the proportion of each sister cultivar by combined genotypes in the sister cultivar group-1. Each color represents a compound genotype, and the thickness of the lines indicates the number of samples. The diagram was produced by the RAWGraphs which is a web-browser based data visualization tool (https://rawgraphs.io/).
그룹-1의 CG1-4와 CG1-5는 전체에서 약 11%와 2%으로 낮은 빈도로 관찰되었으며, 어느 품종내에서도 가장 높은 빈도를 보이지 못했다. 이런 복합유전자형을 가진 춘란 개체는 그룹-1의 다른 품종보다 상대적으로 나중에 채집되어 전파가 덜 되고 새로운 품종으로 등록되지 못했을 가능성이 있다. 그렇지만, CG1-4는 계통도 분석의 dendrogram에서 CG1-2와 아주 가깝게 위치하므로, CG1-2를 가진 개체로부터 후속적으로 발생한 돌연변이로 출현했을 가능성이 있으며, CG1-5 복합유전자형은 계통도와 형매 확률에서 다른 그룹-1의 복합유전자형들과 약간 구분되는 것으로 볼 때, 상대적으로 더 빠른 시기에 분리되었을 가능성이 제기된다.
그룹-2 시료의 대부분을 차지하는 ‘정담’과 ‘미정소’의 경우 비교적 복합유전자형이 품종에 따라 잘 구분되었다. CG2-1의 복합유전자형은 ‘정담’ 시료의 92.3%에서 관찰되었으며, 그 외는 ‘미정소’의 1 시료에서만 관찰되어, CG2-1은 정담-특이적 복합유전자형으로 판단될 수 있다. CG2-2 복합유전자형은 ‘미정소’의 3 시료 중 2 시료에서 관찰되었지만(66.7%), 개체 수가 적기 때문에 CG2-2를 미정소-특이적 복합유전자형으로 여기기는 어렵다고 판단된다. 최근에 경상남도 사천에서 채집된 ‘무명(Anonym)’ 1 개체의 경우는 CG2-2와 동일한 유전자형을 보여, ‘미정소’ 품종에 속하는 것으로 판단된다.
그룹-3의 ‘동천’, ‘채운’ 및 ‘홍귀비’는 춘란 재배자들에게 자매품종으로 알려져 있지 않았지만, 3개 품종의 시료에서 유래한 복합유전자형이 서로 유사성이 높게 관찰되어 본 연구에 포함시켰다. 그룹-3의 품종들에서 유래한 3개의 복합유전자형들은 서로 99.99% 이상의 형매 확률을 보여 가까운 근친의 가능성이 제시되었으며, 계통도 분석의 dendrogram도 역시 서로 매우 가까운 클러스터 형성을 보여주었다. 그럼에도 불구하고 세 품종은 복합유전자형의 분포도로 볼 때 상당히 명확하게 서로 분리되었다. 즉, ‘동광’은 CG3-1 복합유전자형이 92.6%로 대부분을 차지하였으며, 다만 CG3-3 복합유전자형이 7.4% 관찰되었다. CG3-2 복합유전자형은 ‘홍귀비’의 모든 시료(100%)에서 관찰되었고, CG3-3 복합유전자형은 ‘채운’의 모든 시료(100%)에서 관찰되었다. 따라서, CG3-3이 드물게 ‘동광’에서 관찰되기는 하였지만, CG3-1, CG3-2 및 CG3-3은 각각 ‘동광’-, ‘홍귀비’- 및 ‘채운’-특이적 복합유전자형으로 판단된다. 따라서 표현형이 주금소심으로 비슷한 3 품종은 유전적으로 가까운 근친이며, 전라도 지역에서 서로 독립적으로 채집되어 춘란 시장에 등장되었을 가능성이 높다.
그룹-4의 ‘수황’과 ‘살구’도 자매품종으로 알려져 있지 않았지만, 두 품종의 시료에서 유래한 복합유전자형이 서로 유사성이 높게 관찰되어 본 연구에 포함시켰다. 그룹-4에서 관찰된 CG4-3 복합유전자형들은 서로 99.999% 이상의 형매 확률을 보였으며, 계통도에서도 서로 가깝게 위치함을 보여주었다. 그룹-4는 시료 수가 적기 때문에 분포도의 빈도를 계산하는데 적합하지 않지만, ‘수황’의 10 시료에서 CG4-1, CG4-2, CG4-3는 각각 7 시료, 2 시료, 1 시료에서 관찰되었으며, ‘살구’의 2 시료에서는 모두 CG4-2가 관찰되었다. 따라서 ‘수황’과 ‘살구’는 본 연구에서 분석한 12 SSR 좌위의 분석으로는 구분이 어려우며, 서로 시장에 혼재된 것으로 판단된다.
본 연구는 춘란의 자매품종들 간의 유전적 근친성의 정도에 대해 처음으로 분석하였다. 어떤 자매품종 그룹에서 특정 품종-특이적인 복합유전자형이 관찰된 경우도 있었지만 일부 복합유전자형은 여러 품종에서 동시에 관찰되었다. 자매그룹에 따라서는 품종 별 유전적으로 다른 개체들이 서로 혼재되어 있을 가능성이 강하게 제시됨으로써, 앞으로 자매품종들의 정확한 명명을 위해서는 표현형적 특징과 더불어 복합유전자형을 반영한 새로운 기준의 확립이 요구된다. 특히 자매품종 그룹-1에서 ‘홍노을’과 ‘루비’ 및 ‘노을’과 ‘세홍소’와 같이 표현형적 식별과 유전적 식별이 명확하지 않으면서 복합유전자형을 서로 공유하는 자매품종들의 품종 명명의 재정립이 반드시 필요하다고 판단된다. 본 연구는 춘란의 자매품종들 간의 유전적 본질에 대한 이해도를 높이고, 춘란의 자매품종 명명 기준의 재정립 및 거래의 투명성 확보에도 도움이 될 것으로 믿는다.




