서 언
재료 및 방법
잔디 유전자원
잔디의 DNA 추출 및 ITS 기반 CAPS 마커 분석
잔디의 TaqMan SNP genotyping assay 분석
분자생물학적 식별된 한국잔디류의 서식환경별 분포 조사
분자생물학적 식별된 한국잔디 유전자원의 주요 외부형태적 특성인 엽폭 조사
결과 및 고찰
수집된 대량의 한국잔디류 유전자원의 ITS 기반 CAPS 마커 분석과 ITS 기반 TaqMan SNP genotyping assay 분석에 의한 분자생물학적 잔디 식별
분자생물학적으로 식별된 들잔디, 갯잔디 및 교잡종 잔디의 서식환경별 분포 조사
분자생물학적으로 식별된 들잔디, 갯잔디, 들잔디와 갯잔디의 교잡종의 잎의 너비 분석
서 언
잔디(turf grass)는 토양의 침식방지, 도시 녹화, 휴식, 여가생활, 소음 및 공해 방지 등의 목적으로 도로변, 경사면, 산림 훼손지, 건물 정원, 도시 공원, 스포츠 경기장, 골프장, 학교운동장 등의 다양한 장소에 식재되고 있는 원예작물이며 점차 사용 범위가 확대되고 있다(Yu et al., 1974; Choi and Yang, 2006a; Sun et al., 2010; Hyun et al., 2012; Bae et al., 2013; Song et al., 2017). 이러한 잔디는 주로 생육 적온에 따라 고온에 강한 난지형 잔디 및 저온에 강한 한지형 잔디로 크게 분류된다(Kim and Lee, 2010). 난지형 잔디는 들잔디(Zoysia japonica), 갯잔디(Z. sinica), 금잔디(Z. matrella), 왕잔디(Z. macrostachya), 버뮤다그라스(Cynodon dactylon) 등이 있고, 한지형 잔디는 켄터키 블루그라스(Poa pratensis), 크리핑 벤트그라스(Agrostis stolonifera), 라이그라스(Lolium perenne), 톨훼스큐(Festuca arundinacea) 등이 있다(Kim and Lee, 2010; Bae et al., 2013; Choi, 2017). 우리나라에서 주로 자생하고 있는 잔디는 난지형 잔디로 화본과 Zoysia 속의 한국잔디류(Zoysiagrass)이며, 주로 들잔디 및 갯잔디가 많이 자생하고 있고, 드물게 금잔디 및 왕잔디가 자생하고 있다(Yu et al., 1974; Li et al., 2006; Bae et al., 2010; Kim and Lee, 2010; Sun et al., 2010; Hyun et al., 2012; Bae et al., 2013; Chung et al., 2013; Lee et al., 2013; Choi, 2017). 이러한 한국잔디류는 주로 영양번식하는 다년생의 타가수정작물이며, 온대기후부터 열대기후의 아시아 지역인 한국 및 중국, 일본, 뉴질랜드, 네팔, 태국, 필리핀 등의 아시아 지역에 자생하는 대표적인 난지형 잔디이다(Yu et al., 1974; Kim and Lee, 2010; Sun et al., 2010; Hyun et al., 2012). 난지형 잔디인 한국잔디류는 한지형 잔디보다 고온 및 건조, 해안지대의 염분, 병충해, 답압 등에 강한 특성이 있어 우수한 신품종 육종을 위한 중요한 소재로 사용되는 유전자원이다(Yu et al., 1974; Li et al., 2006; Sun et al., 2010; Hyun et al., 2012; Chung et al., 2013). 한국잔디류에 속하는 들잔디는 주로 산, 내륙, 해안가에서 자생하며 갯잔디는 갯벌, 바닷가의 바닷물 인접지역에서 자생하기 때문에 들잔디에 비해 갯잔디가 내염성은 더 강하다(Yu et al., 1974; Hong and Yeam, 1985; Li et al., 2006; Sun et al., 2010; Hyun et al., 2012; Chung et al., 2013). 들잔디와 갯잔디의 각 생육특성을 살펴보면, 들잔디의 경우에는 지표면 근쳐에서 낮게 자라는 low-growing creeping type이며 갯잔디의 경우에는 지표면 위로 약간 서서 자라는 high-growing type이고, 들잔디에 비해 갯잔디의 생육속도는 매우 느린편이다(Hong and Yeam, 1985). 이러한 생육특성으로 잔디 잎의 밀도가 달라지는데, 본 연구소의 들잔디와 갯잔디의 교잡종의 경우에는 갯잔디보다 밀도가 확연히 높은 타입이 많아 고밀도의 내염성 품종으로 더 우수한 품종을 육성할 수 있어 교잡종도 중요한 유전자원이다.
유전자원에 대한 접근 및 그 이용으로부터 발생하는 이익의 공정하고 공평한 공유에 관한 생물다양성에 관한 협약 나고야 의정서에 따라 우리나라에서도 국내의 다양한 유전자원을 대상으로 나고야 의정서의 적용과 관련된 대비가 시급하다. 우리나라는 전국에 자생하는 잠재된 우수한 한국잔디류 유전자원을 보유하고 있으나 아직 국내 유전자원을 수집 및 보존, 관리, 분류 동정, 품종 육성, 산업화가 해외에 비해 적은 실정이다(Choi et al., 1997; Choi and Yang, 2004, 2006a, 2006b; Yang et al., 2016a; Choi, 2017; Song et al., 2017; Choi et al., 2018; Yang et al., 2021a, 2021b). 본 연구팀은 2012년부터 최근까지 한국에 자생하는 한국잔디류를 각각 다른 지역에서 수집하여 제주대학교 아열대원예산업연구소 유전자원 시험포장 및 비닐온실에서 보존, 증식, 분류 및 관리하고 있으며, 선발 및 돌연변이 육종, 품종보호 출원 및 등록하여 우수한 형질의 한국잔디류를 육성해 왔다(Yang et al., 2016a; Hong et al., 2017; Song et al., 2017; Yang et al., 2021a, 2021b). 또한, 한국에 많이 자생하고 있는 들잔디와 갯잔디의 난지형 한국잔디류와 켄터키블루그라스와 크리핑벤트그라스의 한지형 잔디들을 신속 정확하게 분자생물학적으로 종별 식별할 수 있는 분자마커를 개발하였다(Hong et al., 2017). 다양한 장소에서 수집 보존중인 한국잔디류의 경우, 확연히 다른 환경인 산에서 수집된 들잔디나 갯벌 또는 바닷가 바위에서 수집된 갯잔디들은 외부형태학적인 명확한 차이 때문에 종 분류 동정은 쉽게 가능하다. 그러나, 해안가와 섬지역 같은 바다와 가까운 환경에서 수집된 야생 들잔디, 갯잔디 및 들잔디와 갯잔디의 교잡종은 외형이 매우 유사하여 비전문가의 경우 외부형태학적 분류동정이 쉽지 않다(Yang et al., 1995; Hong et al., 2017). 이러한 이유로, 본 연구실에서는 한국잔디류의 DNA 바코드 구간인 nuclear ribosomal DNA(nrDNA)의 internal transcribed spacer(ITS) 염기서열 차이에 의한 cleaved amplified polymorphic sequence(CAPS) 마커를 개발하여 들잔디, 갯잔디, 그들의 교잡종의 분류 동정 기법을 성공적으로 확립하였다(Hong et al., 2017). ITS 기반 DNA 바코드 분석은 다양한 생물 분류 동정을 위하여 활발히 연구되어 왔으며, 종 및 속간 유연관계, 진화 양상, 한약제 위품 구별 등 신속 정확한 분석을 위하여 활용되고 있다(Gehrig et al., 2001; Hebert et al., 2003; Kunihisa et al., 2003; Kress et al., 2005; Han et al., 2006; Kress and Erickson, 2008; Baigalmaa et al., 2009; CBOL Plant Working Group, 2009; Ahn et al., 2010; Gao et al., 2010; Koch, 2010; Yao et al., 2010; China Plant BOL Group, 2011; Hong et al., 2012; Kim et al., 2012; Moon et al., 2013; Kim et al., 2015; Hong et al., 2017).
수집된 야생 한국잔디류 유전자원은 본 연구팀에서 개발한 ITS 염기서열 차이 기반 CAPS 분자마커로 들잔디, 갯잔디, 그들의 교잡종까지 정확하게 식별이 가능하였지만, 다량 분석을 수행하는데 많은 시간과 노동력이 소요되어, 좀 더 신속하고 정확한 식별방법이 요구되었다. 최근 형광물질을 활용한 single-nucleotide polymorphism(SNP) 기반 TaqMan probe 및 Kompetitive allele-specific PCR(KASP)기술 등의 방법으로 단일염기다형성 유전자형 분석(SNP genotyping assay)을 손쉽게 할 수 있는 분자표지 기술이 개발되어 활용되고 있다. TaqMan probe에 의한 SNP genotyping assay 분석법은 원하는 염기서열 구간의 SNP를 Real-time PCR을 이용하여 유전변이, 생물종 식별 및 작물 육종시 우수라인 선발 등의 분석에 활용되고 있다(Tan et al., 2017; Broccanello et al., 2018; Ayalew et al., 2019; Kang et al., 2020; Shin et al., 2020). ITS 기반 CAPS 마커는 정확도는 높으나 많은 시간과 노동력이 소요된다. TaqMan probe 기술의 SNP genotyping assay set 분석법은 Real-time PCR 후 분류 동정 결과를 그래프 및 표로 신속 정확하게 확인할 수 있어 시간과 노동력을 줄일 수 있다.
본 연구에서는 기존 ITS 기반 CAPS 마커를 활용하여 수집되어 보존 중인 다량의 한국잔디류 유전자원을 추가 분류 동정하였다. 또한, 다량의 유전자원을 좀 더 신속 정확하게 분류 동정 하기 위하여 ITS 기반 TaqMan SNP genotyping assay 분석법을 개발하여 종을 구별하였으며, ITS 기반 CAPS 마커의 결과와 비교하였다. 또한 분류 동정된 들잔디, 갯잔디, 교잡종을 기반으로 서식환경별 분포를 조사하였으며, 기존 외부형태학적으로 주요한 분류 형질인 잎의 너비(엽폭)가 들잔디와 갯잔디 교잡종의 분류동정에 이용할 수 있는지 분석하였다.
재료 및 방법
잔디 유전자원
본 연구에서 사용된 한국잔디류 유전자원은 본 연구팀이 2012년부터 2018년도까지 한국 주요 지역인 서울 및 인천을 포함하는 경기도권, 강원도, 충청남북도, 전라남북도, 경상남북도, 제주도 지역을 중심으로 수집하였다. 특히 한국잔디류의 자생지인 각 지역의 내륙의 국립공원 산 및 동 ․ 서 ․ 남해안 부근의 해안과 섬, 제주 지역의 오름 및 해안과 부속 섬 등의 각각 다른 지역에서 한국잔디류를 10–15cm 내외 사각형으로 수집하였고, 국립공원으로 명시된 곳은 국립공원관리공단의 허가하에 수집하였다. 수집된 영양체는 제주대학교 아열대원예산업연구소에서 시험포장 및 비닐온실에서 보존 및 증식하였고, 수집된 종자는 저온저장고에 보존하였다. 수집하여 보존된 한국잔디류는 620점(영양체 555점 및 종자 65점)이며, 영양체의 각 잔디의 잎은 엽폭을 조사한 후에 에탄올 소독하여 DNA 추출 재료로 사용되었다. 분자생물학적 분류 동정을 위하여 사용된 대조구는 기존에 본 연구팀에서 보고한 ITS 기반 CAPS 마커방법(Hong et al., 2017)으로 분자생물학적 분류동정이 이미 완료된 들잔디(Z. japonica, China; Zj-C)의 판매 종자 및 수집된 갯잔디(Z. sinica, Jeju, Korea; Zs-C)종자, 들잔디와 갯잔디의 교잡종(Z. japonica and Z. sinica hybrid, Jeju, Korea; H-C) 종자로부터 확보한 잔디이며, ITS 기반 CAPS 마커 및 ITS 기반 TaqMan SNP genotyping assay 분석법에 의한 분자생물학적 분류를 위해서 대조구로 사용되었다(Table 1). 대조구인 종자는 소독 및 파종 후 MS(Murashige and Skoog, 1962) 기본 배지에 배양되었으며, 각 잔디 잎은 DNA 추출 재료로 사용되었다. 수집하여 보존 및 증식한 자생 Zoysia 속 한국잔디류 들잔디 및 갯잔디 계통의 영양체 중에서 450점은 대조구와 비교 분석하여 ITS 기반 CAPS 마커 및 ITS 기반 TaqMan SNP genotyping assay의 대량 분류 동정을 위한 DNA 추출 재료로 사용되었다(Table 1). 분자생물학적 분류 동정된 들잔디, 갯잔디 및 들잔디와 갯잔디의 교잡종은 서식환경별 분포를 분석하였다.
Table 1.
Molecular identification of collected zoysiagrass plants: A total of 450 collected zoysiagrass plants were classified into 234 Zoysia japonica plants, 62 Zoysia sinica plants, and 154 Z. japonica and Z. sinica hybrid plants by molecular identification using the ITS-based CAPS marker analysis and ITS-based TaqMan SNP genotyping assays
잔디의 DNA 추출 및 ITS 기반 CAPS 마커 분석
분자생물학적 분류동정을 위하여 각 잔디로부터 DNA 추출은 GeneAll 회사의 ExgeneTM Plant SV Mini Kit(GeneAll, Korea)를 이용하여 추출하였으며, 추출한 DNA는 0.8% 겔의 전기영동으로 확인한 후 정량하여 PCR에 사용하였다. 이후 과정은 본 연구실에서 개발한 ITS 기반 CAPS 마커 분석(Hong et al., 2017)을 이용하였으나, 본 연구에서는 기존에 사용된 제한효소 중에서 가장 효율이 좋았던 Fau I를 사용하여 ITS 기반 CAPS 마커 분석되었으며, 간단한 방법은 다음과 같다. ITS 증폭을 위한 primer는 Gehrig et al.(2001)이 보고한 ITS1-forward primer(5’-TCCGTAGGTGAACCTGCGG-3’)와 ITS4-reverse primer(5’-TCCTCCGCTTATTGATATGC-3’)가 사용되었다. PCR은 AccuPower® PCR PreMix(Bioneer, Korea) 및 PCR Thermal Cycler Dice(TaKaRa, Japan)를 이용하였으며 95℃에서 5분간 전처리 후, 95°C에서 30초, 57°C에서 30초, 72°C에서 40초를 30회 반복한 후 72°C에서 10분간 후처리 하였다. 증폭된 686bp 및 687bp의 PCR 산물은 ExpinTM PCR SV Kit(GeneAll, Korea)를 이용하여 정제되였으며, 정제된 PCR 산물과 5 unit의 Fau I 제한효소(NEB, England), 10X CutSmart buffer가 포함된 14µL 반응액은 최적 반응 온도에서 충분한 시간 동안 반응시켰다. 최종 반응물은 2% agarose gel에서 전기영동 하여 CAPS marker 분석하였으며, 갯잔디(Zs)의 경우에 480bp, 들잔디(Zj)의 경우에 653bp, 들잔디와 갯잔디의 교잡종의 경우에 480bp, 653bp 부근에서 전기영동 주요 밴드를 확인하여 CAPS marker를 분석하여 분류 동정하였다.
잔디의 TaqMan SNP genotyping assay 분석
각 잔디로부터 추출된 DNA는 TaqMan SNP genotyping assay 분석을 위한 재료로 사용되었다. TaqMan SNP Genotyping Assays를 수행하기 위하여 SNP는 대조구의 들잔디 및 갯잔디간의 ITS 염기서열의 567bp에 위치한 SNP(A 또는 G)의 유전자형을 Real-time PCR에 의해 확인하기 위하여 primer 와 probe로 구성된 SNP 유전자형 분석 세트(TaqMan Genotyping Master Mix)인 Forward primer(ACGGCACATGTTGTTCTTGGA)와 Reverse primer(CCTGAGGGCCATCATGTCA), Reporter 1 dye(VIC), Reporter 1 Sequence(TTGTGACCTGCATCGTAA), Reporter 1 Quencher(NFQ), Reporter 2 dye(FAM), Reporter 2 Sequence(TGTGACCTGCGTCGTAA), Reporter 2 Quencher(NFQ)를 제조하였다(TaqMan Genotyping Master Mix, Applied Biosystems, USA). 한국잔디류 유전자원들 각각 5–20ng/µL genomic DNA 1µL와 최종 1 X TaqMan Genotyping Master Mix(Applied Biosystems, USA), 1 X GoTaq Probe qPCR Master Mix(Promega, USA)를 혼합하여 96-well plate에 넣은 후 LightCycler 480 Real time PCR System(Roche, Swiss)기기를 이용하여 95°C 10분 1회, 95°C 15초 및 60°C 1분 40회 반복으로 LightCycler 480 Real time PCR System 기기의 분석프로그램 중에서 Endpoint Genotyping으로 TaqMan SNP Genotyping Assays 분석을 수행하였다. 대조구는 ITS 기반 CAPS 마커방법(Hong et al., 2017)으로 분자생물학적 분류동정이 이미 완료된 들잔디(Zj-C)의 판매 종자 및 수집된 갯잔디(Zs-C)종자, 들잔디와 갯잔디의 교잡종(H-C) 종자로부터 확보한 잔디를 분자생물학적 분류 동정의 대조구로 사용되었다(Fig. 1B and Table 1).
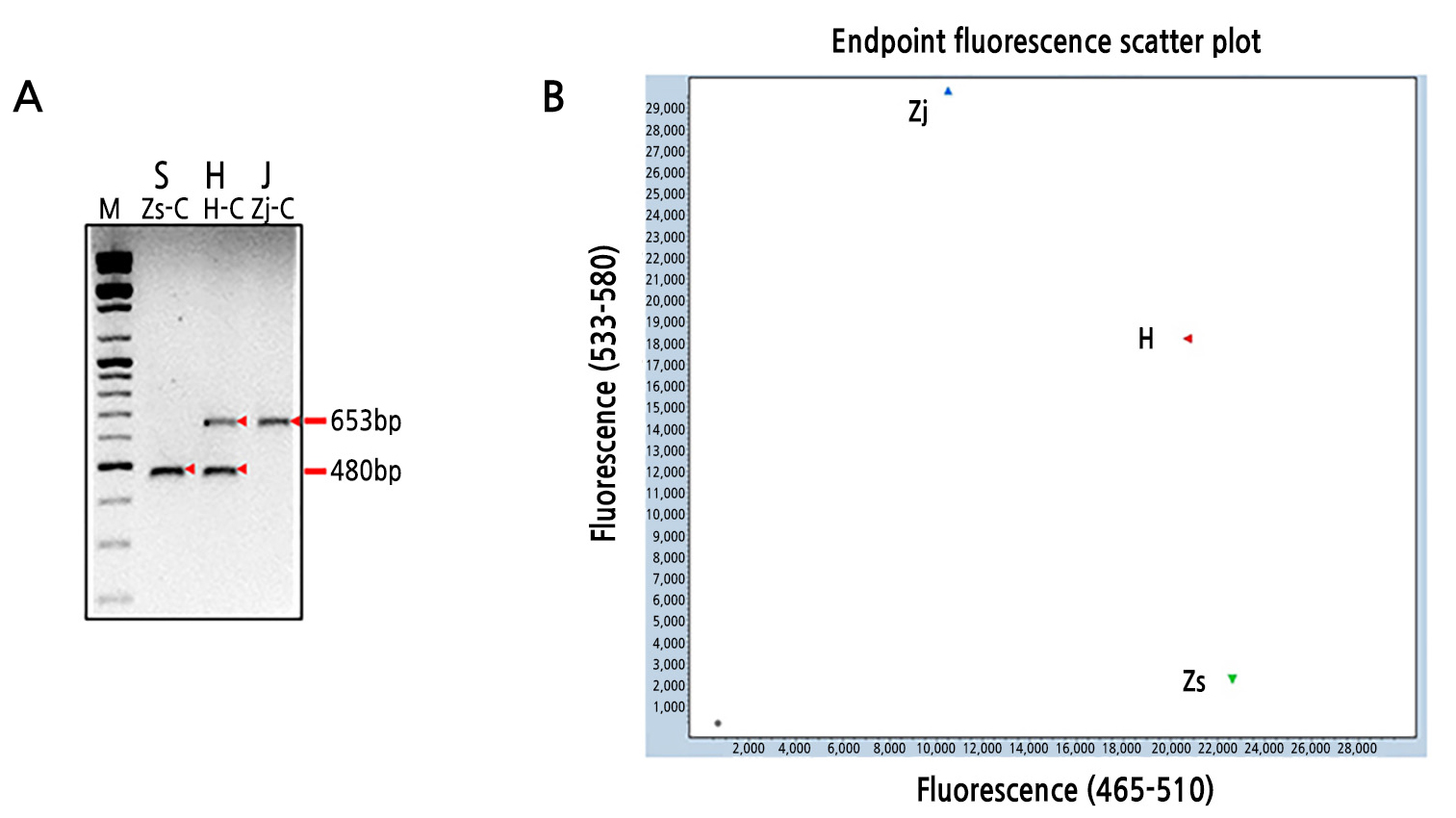
Fig. 1.
Molecular identification of control zoysiagrass plants using ITS-based CAPS marker analysis (A), ITS-based TaqMan SNP genotyping assay (B): Analysis of the ITS-based CAPS marker and ITS-based TaqMan SNP genotyping assay allowed the classification of control zoysiagrass plants into Z. japonica plants (Zj-C, Zj), Z. sinica plants (Zs-C, Zs), and Z. japonica and Z. sinica hybrid plants (H-C, H); (A), ITS PCR products were purified and digested by Fau I; M is the size marker. (B), a three-cluster allelic discrimination plot generated with the TaqMan SNP genotyping assay.
분자생물학적 식별된 한국잔디류의 서식환경별 분포 조사
분자생물학적 분류 동정된 한국잔디류 들잔디, 갯잔디 및 들잔디와 갯잔디의 교잡종에 대한 각 서식환경별 분포를 조사하였다. 분류 동정된 한국잔디류 450개체는 종별(들잔디, 갯잔디 및 들잔디와 갯잔디의 교잡종)로 분류하였고, 그 후에는 고지대(국립공원 산 및 오름) 및 저지대(내륙, 해안 및 섬)로 분류하여 각 종별 한국잔디류의 서식환경별 분포를 조사하였다.
한국잔디류 450개체의 지역은 강원도(오대산, 설악산, 치악산)의 산 3개, 경기도권(북한산, 백령도, 강화도, 영종도, 영흥도, 덕적도)의 산 1개 및 섬 5개, 충청북도(속리산, 소백산, 월악산)의 산 3개, 충청남도(계룡산, 안면도, 외연도)의 산 1개 및 섬 2개, 전라북도(지리산, 덕유산, 내장산, 고창, 선유도)의 산 3개, 내륙 및 해안가 1개, 섬 1개, 전라남도(월출산, 순천, 진도, 흑산도, 보길도, 안도, 금오도, 거금도, 고금도, 압해도, 자은도, 암태도, 안좌도, 팔금도, 비금도, 도초도)의 산 1개 및 해안 1개, 섬 14개, 경상북도(주왕산, 가야산, 울릉도)의 산 2개 및 섬 1개, 경상남도(창선도, 거제도)의 섬 2개, 제주도(신례, 효돈, 영평, 가시리, 안덕, 한라산 관음사, 한라산 성판악, 한라산 돈내코, 한라산 영실, 이승악(이승이오름), 대록산(큰사슴이오름), 거문오름, 백약이오름, 온평, 신양, 성산, 위미, 보목, 모슬포, 고산, 대정, 표선, 한림, 협제, 하도리, 도두, 가파도, 비양도, 추자도)의 내륙 5개, 산 및 오름 5개, 해안가 13개, 섬 3개이다(Table 1).
분자생물학적 식별된 한국잔디 유전자원의 주요 외부형태적 특성인 엽폭 조사
한국잔디류의 외부형태학적으로 주요한 분류 형질인 잎의 너비(엽폭)는 들잔디, 갯잔디의 분류동정의 주요 형질로 이용되었다(Yang et al., 1995). 엽폭은 전자 자를 이용하여 3 반복으로 측정하였으며, 각 잔디 잎은 DNA 추출 재료로 사용되어 분자생물학적 ITS 기반 CAPS 마커 및 ITS 기반 TaqMan SNP genotyping assay 방법에 의하여 분류 동정되었다. 최종 분자생물학적으로 분류 동정된 들잔디, 갯잔디, 들잔디와 갯잔디의 교잡종은 외부형태학적 분류방법인 엽폭과 교잡종과의 상관관계를 비교 분석하였다.
결과 및 고찰
수집된 대량의 한국잔디류 유전자원의 ITS 기반 CAPS 마커 분석과 ITS 기반 TaqMan SNP genotyping assay 분석에 의한 분자생물학적 잔디 식별
본 연구팀은 기존에 한국잔디류의 들잔디 및 갯잔디, 서양잔디류의 켄터키블루그라스 및 벤트그라스의 신속 정확하게 식별하기 위하여 분자생물학적 분류동정법으로 ITS 기반 DNA 바코드 분석 및 ITS 기반 CAPS 마커 분석(Hong et al., 2017)을 개발하였으며, 특히 해안가에서 자생하는 외부형태가 비슷하여 쉽게 식별이 어려웠던 한국잔디류의 들잔디 및 갯잔디, 들잔디와 갯잔디의 교잡종에서 ITS 기반 CAPS 마커 분석에 의하여 전기영동상 DNA 밴드 크기 차이로 정확한 분류 동정법을 개발하였다(Hong et al., 2017). 본 연구에서는 개발된 ITS 기반 CAPS 마커 분석을 활용하여 한국 전역(서울 및 인천을 포함하는 경기도권, 강원도, 충청남북도, 전라남북도, 경상남북도 지역의 내륙, 국립공원 산, 동 〮 서 〮 남해안 부근의 해안과 섬, 제주 지역의 오름 및 해안과 부속 섬 등)에서 다량 수집된 한국잔디류 영양체를 추가 분류동정 하였다(Fig. 1A). 수집된 한국잔디류 영양체 432개를 ITS 기반 CPAS 마커 분석법으로 분자생물학적 분류동정한 결과, 총 432개의 한국잔디류가 들잔디(Zj, J) 228개와 갯잔디(Zs, S) 52개, 들잔디와 갯잔디의 교잡종(H) 151개로 분류 동정되었다(Table 1, Figs. 1A and Fig. 2). 기존의 보고에서 ITS 기반 CPAS 마커 분석법 중에서 제한효소 Fau I으로 들잔디와 갯잔디, 교잡종을 분류 동정 수행할 경우, 2% agarose gel 전기영동상에서 들잔디의 경우에는 653bp와 33bp 중에서 653bp 크기의 절단된 DNA 조각의 주요 밴드 한 개, 갯잔디의 경우에는 480bp와 91bp 중에서 480bp 크기의 주요 밴드 한 개, 들잔디와 갯잔의 교잡종은 480bp, 653bp 부근에서 주요 밴드 두 개를 확인하여 분류 동정되었다(Hong et al., 2017)(Fig. 1A). 본 연구에서도 다량의 한국잔디류 영양체 432개의 ITS 기반 CAPS 마커 분석 결과 653bp의 들잔디(J), 480bp의 갯잔디(S), 480bp와 653bp의 들잔디와 갯잔디의 교잡종(H)으로 각각 절단된 DNA 주요 밴드가 확인되어 각각 들잔디, 갯잔디, 교잡종으로 추가 분류 동정되었다(Fig. 2).
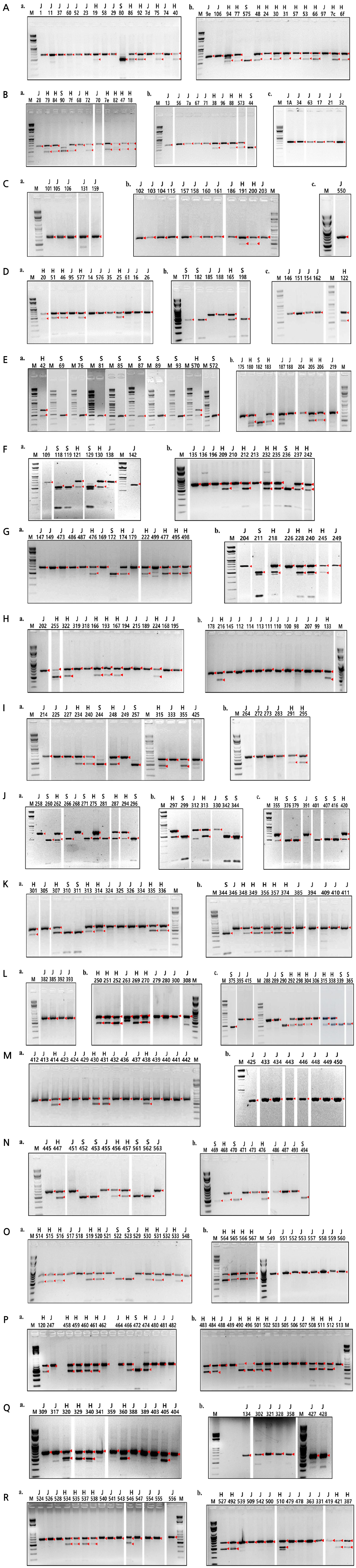
Fig. 2.
Molecular identification of collected zoysiagrass plants using the ITS-based CAPS marker analysis: Analysis of the ITS-based CAPS marker allowed the classification of the collected zoysiagrass plants into Z. japonica plants (J), Z. sinica plants (S), and Z. japonica and Z. sinica hybrid plants (H); M is the size marker; sample numbers are shown in Table 1.
ITS 기반 CAPS 마커 분석 방법은 들잔디, 갯잔디, 그들의 교잡종인 들잔디와 갯잔디를 정확하게 식별하였으나 대량 분석을 수행하는데 많은 시간과 노동력이 소요되었기 때문에, 본 연구에서는 좀 더 신속한 분자생물학적 분류 동정법인 ITS 기반 TaqMan SNP genotyping assay 분석 방법을 개발하여 대량 한국잔디류의 분류 동정을 수행하였다. ITS 기반 TaqMan SNP genotyping assay 분석에 의하여 대조구인 들잔디, 갯잔디, 들잔디와 갯잔디의 교잡종을 분류 동정한 결과, 대조구의 경우 들잔디(Zj)는 Y축에 가까이에 삼각형 모양의 점이 위치하였으며, 갯잔디(Zs)의 경우 X축, 들잔디와 갯잔디의 교잡종(H)의 경우 X축과 Y축 사이 45도 각도로 위치하여 분류 동정됨을 확인할 수 있었다(Fig. 1B). 이러한 결과를 바탕으로 대조구 및 대량 수집 한국잔디류 영양체 234개를 ITS 기반의 TaqMan SNP genotyping assay 분석한 결과, 총 234개의 한국잔디류가 들잔디(Zj) 124개와 갯잔디(Zs) 30개, 들잔디와 갯잔디의 교잡종(H) 80개로 분류 동정되었다(Tables 1, 2 and Fig. 3). 한국잔디류 영양체 234개 중에서 216개는 ITS 기반 CAPS 마커 분석 및 ITS 기반 TaqMan SNP genotyping assay 분석의 두 가지 방법으로 분류 동정되었으며, 그 결과는 두 가지 방법 모두 일치된 결과로 확인되어 본 연구에서 개발된 ITS 기반 TaqMan SNP genotyping assay 방법으로 들잔디(Zj), 갯잔디(Zs), 들잔디와 갯잔디의 교잡종(H)을 신속 정확하게 식별 가능함을 알 수 있었다(Table 1).
추가로 일부 잔디 제주 표선에서 수집된 125번, 전라남도 압해도 230번, 231번, 233번, 239번, 253번, 전라남도 암태도 274번, 276번, 전라남도 안좌도 284번, 285번, 286번, 전라남도 팔금도 296번, 전라북도 선유도 381, 제주 추자도 390번, 경상남도 창선도 406번, 경상남도 거제도 418번, 전라북도 내장산 446번, 경기도권 백령도 545번의 총 18개는 TaqMan SNP genotyping assay 분석만을 수행하여 신속하게 각각 들잔디 6개, 갯잔디 10개, 교잡종 2개로 분류 동정 되었다(Table 1, 2 and Fig. 3).
이러한 TaqMan probe 기술의 SNP genotyping assay 방법은 형광물질을 활용하여 genomic DNA 추출 및 TaqMan probe 이용한 ITS의 SNP 구간을 중심으로 Real-time PCR 증폭 후 전기영동 없이 컴퓨터 화면에서 그래프 및 표로 신속하게 종 분류 동정을 확인 할 수 있으며, 최근 SNP genotyping assay 기술은 생물종 식별, 유전변이 유무, 작물 육종과정에서 우수 계통의 선발 등의 중요한 분자표지 기술로 개발 및 활용되고 있다(Tan et al., 2017; Broccanello et al., 2018; Ayalew et al., 2019; Kang et al., 2020; Shin et al., 2020).
본 연구에서도 한국잔디류 들잔디, 갯잔디, 들잔디와 갯잔디의 교잡종 분류 동정 결과가 ITS 기반 CAPS 마커 분석과 ITS 기반의 TaqMan SNP genotyping assay 분석에서 결과가 일치하였으며, ITS 기반의 TaqMan SNP genotyping assay 분석법은 ITS 기반 CAPS 마커 분석법보다 신속하게 식별 가능함이 확인되었다.
분자생물학적으로 식별된 들잔디, 갯잔디 및 교잡종 잔디의 서식환경별 분포 조사
ITS 기반 CAPS 마커 분석 및 ITS 기반 TaqMan SNP genotyping assay 분석을 이용하여 2012년부터 2018년도까지 한국 주요 지역의 각각 다른 위치에서 수집한 자생한국잔디류 약 620점(영양체 555점 및 종자 65점) 중 영양체 450점(개체)을 분석한 결과, 들잔디(Zj) 234개, 갯잔디(Zs) 62개 및 들잔디와 갯잔디의 교잡종(H) 154개로 분류 되었다(Tables 1, 2, and Figs. 1, 2, 3).
본 연구에서 분자생물학적으로 들잔디, 갯잔디 및 들잔디와 갯잔디의 교잡종으로 분류 동정된 잔디의 서식환경을 분석한 결과, 들잔디는 산 및 오름에서 총 71 개체 그리고 내륙 및 해안가 부근, 섬지역에서 총 135개체의 자생 들잔디가 분포하고 있음을 알 수 있었다(Table 1). 갯잔디는 주로 바닷물이 인접한 해수면 인근의 바위지대에서 분포하고 있으나, 고지대인 산 및 오름에는 갯잔디는 분포하지 않았다.
Table 2.
Molecular Identification of collected zoysiagrass plants using a three-cluster allelic discrimination plot generated with the TaqMan SNP Genotyping Assay: Molecular classification by endpoint fluorescence of Z. japonica plants (Zj), Z. sinica plants (Zs), and Z. japonica and Z. sinica hybrid plants (H). FAM or VIC dyes detect samples that are homozygous for allele X or allele Y, respectively; Allele X, allele Y, and both alleles indicate Zs, Zj, and H, respectively
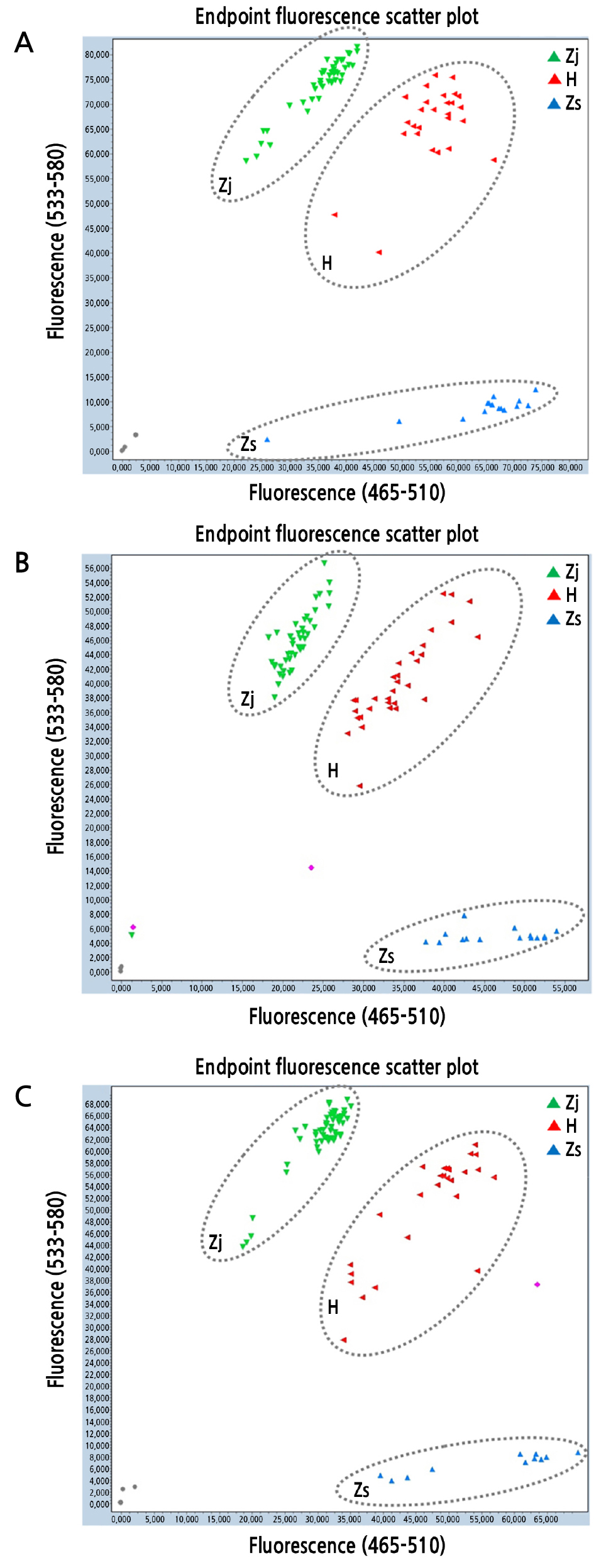
Fig. 3.
Molecular identification of collected zoysiagrass plants using a three-cluster allelic discrimination plot generated with the TaqMan SNP genotyping assay: Molecular classification of 234 samples of zoysiagrass plants (respective group A ~ C) as shown in Table 2 by endpoint fluorescence scatter plots (group A ~ C) of Z. japonica plants (Zj), Z. sinica plants (Zs), and Z. japonica and Z. sinica hybrid plants (H).
갯잔디는 저지대 중에서 섬지역 해안가 바닷물이 인접한 부근인 경기도권(백령도, 강화도, 덕적도)에서 7개, 충남(안면도)에서 2개체, 전라북도(선유도)에서 3개체, 전라남도(보길도, 거금도, 압해도, 자은도, 암태도, 안좌도, 팔금도, 비금도, 도초도, 진도)에서 30개체, 경상남도(거제도, 창선도) 3개체, 그리고 제주도(제주도 해안가 지역들, 비양도)에서 17개체가 수집되어 자생 갯잔디가 소량 분포하고 있었다(Table 1). 반면에 일부 섬지역인 경기도권의 영종도 및 영흥도, 충청남도 외연도, 전라남도 흑산도 및 안도, 금오도, 고금도, 경상북도 울릉도, 제주 가파도 및 추자도에서 본 연구팀이 외부형태적으로 갯잔디로 예상하여 수집했지만 모두 들잔디와 갯잔디의 교잡종으로 최종 분자생물학적 분류 동정되어 분자생물학적 갯잔디로 분류 동정된 잔디는 없었다(Table 1).
들잔디와 갯잔디의 교잡종은 아주 드물게 고지대인 소백산 및 계룡산, 지리산, 한라산 영실에서 총 4개체, 주왕산에서 총 2개체가 일부 인공적으로 조경이 되어있는 국립공원관리공단의 일부 탐방안내소, 등산로 및 절의 경내 인근에 확인되었다(Table 1). 또한, 수집된 모든 섬 지역에서 자생 들잔디와 갯잔디의 교잡종이 총 114개체가 확인되었고, 국립공원 산 보다는 내륙 및 해안가, 섬지역에 걸쳐 다양한 장소에 분포하고 있음을 알 수 있었다(Table 1). 한국잔디류의 FT-IR 대사체 분석에 의하여 식별된 들잔디 및 갯잔디 계통에서 들잔디는 고지대 국립공원 산에서, 갯잔디는 저지대 해안가에서 주로 분포하였으며(Yang et al., 2016b), 들잔디는 내륙부터 해안까지 넓은 범위로 분포되어 있으나 갯잔디와 그들의 교잡종은 서해안 및 남해안, 도서지역 부근에 분포하고 있다고 보고되었다(Choi, 2017).
한국잔디류는 주로 타가수정하여 가까운 거리에서 분포하고 있는 들잔디 및 갯잔디의 경우 자연적으로 종간 교잡되기 때문에(Choi et al., 2008; Choi, 2017), 갯잔디가 자생하지 않는 고지대의 국립공원 산 및 오름 보다는 저지대의 해안가 및 내륙에서 들잔디와 갯잔디가 자연적으로 종간 교잡된 것으로 추정된다. 본 연구에서도 분자생물학적으로 분류 동정한 수집된 들잔디는 주로 국립공원 산 및 오름, 내륙, 해안가 부근에서 분포되었고, 갯잔디는 바닷물에 자주 잠기는 바위나 돌, 해안가의 바닷물이 인접한 부근에서 분포되었으며, 들잔디와 갯잔디의 교잡종은 국립공원 산 및 오름보다 내륙 및 해안가, 섬지역에 걸쳐 다양한 장소에 분포되어 기존의 보고와 유사하였다.
분자생물학적으로 식별된 들잔디, 갯잔디, 들잔디와 갯잔디의 교잡종의 잎의 너비 분석
들잔디 및 갯잔디 등 한국잔디류의 외부형태적 주요 분류동정법으로는 육안으로 쉽게 관찰 가능한 엽폭 크기 등이 활용되고 있다(Hong and Yeam, 1985; Yang et al., 1995; Choi et al., 1997; Bae et al., 2010; Lee et al., 2013; Yang et al., 2021a, 2021b). 최종 분자생물학적으로 분류 동정된 들잔디, 갯잔디, 들잔디와 갯잔디의 교잡종을 대상으로 기존 외부형태학적으로 주요한 분류 형질인 엽폭이 교잡종의 분류동정에 이용할 수 있는지 분석하였다. 분자생물학적 분류 동정된 들잔디(Zj) 234개, 갯잔디(Zs) 62개, 들잔디와 갯잔디의 교잡종(H) 154개의 한국잔디류 총 450개의 각 종별 엽폭 평균은 들잔디의 경우 4.52 ± 0.05mm, 갯잔디의 경우 2.97 ± 0.07mm, 들잔디와 갯잔디의 교잡종의 경우 3.51 ± 0.05mm로 분석되었다(Table 3). 들잔디의 엽폭 평균은 갯잔디보다 약 1.5배 넓고 들잔디와 갯잔디의 교잡종 보다 약 1.3배 넓게 분석되었으며, 갯잔디는 들잔디와 갯잔디의 교잡종 보다 약 1.2배 좁게 분석되어, 들잔디와 갯잔디의 교잡종은 들잔디와 갯잔디의 중간값을 보였다(Table 3).
Table 3.
Leaf blade width as a main external morphological classification characteristic of the molecularly identified Z. japonica plants (Zj), Z. sinica plants (Zs), and Z. japonica and Z. sinica hybrid plants (H)
| Sample Species | Leaf blade width (mm) |
| Z. japonica (Zj) | 4.52 ± 0.05 |
| Z. japonica and Z. sinica hybrid (H) | 3.51 ± 0.05 |
| Z. sinica (Zs) | 2.97 ± 0.07 |
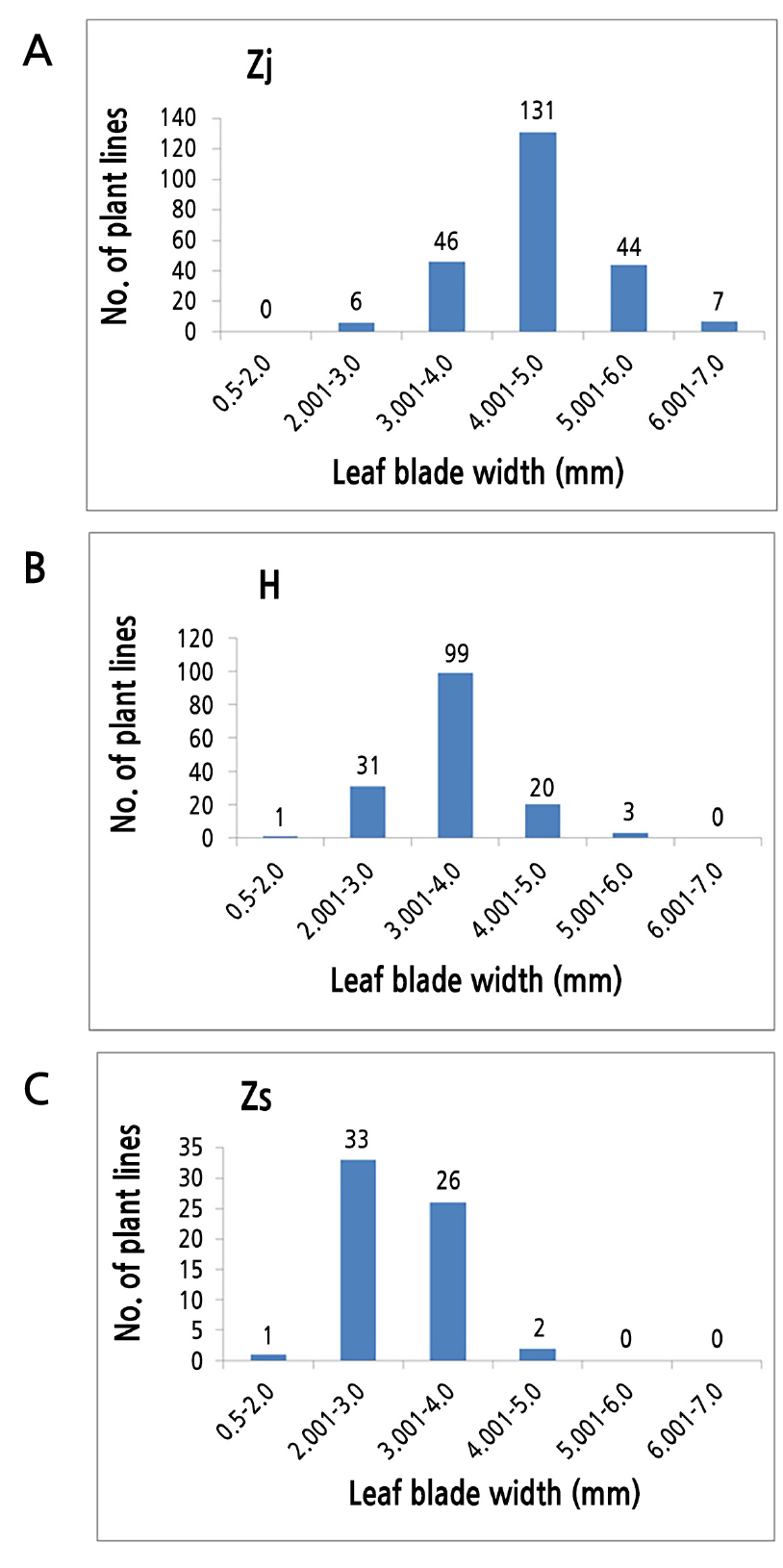
각 종별로 엽폭 크기의 범위를 조사하기 위하여, 총 450개체의 한국잔디류는 각 종에서 엽폭 크기(0.5–2.0, 2.001–3.0, 3.001–4.0, 4.001–5.0, 5.001–6.0, 6.001–7.0mm)별로 잔디 개수를 확인하였다. 그 결과, 들잔디의 경우에는 2.001–7.0mm로 다양한 크기의 엽폭을 확인 할 수 있었으며 특히 들잔디(Zj) 234개체 중에서 131개체의 엽폭 크기가 4.001–5.0mm로 가장 많았으며, 2.001–3.0mm와 6.001–7.0mm의 13개체를 제외한 대부분의 들잔디 221개체의 엽폭 크기가 3.001–6.0mm의 범위임을 알 수 있었다(Fig. 4).
갯잔디(Zs) 62개체의 엽폭 크기는 0.5–5.0mm의 다양한 범위로 2.001–3.0mm에서 33개체, 3.001–4.0mm에서 26개체이며, 대체로 갯잔디의 엽폭 크기가 2.001–4.0mm의 범위로 들잔디 보다 작은 엽폭 크기의 범위임을 알 수 있었다(Fig. 4).
들잔디와 갯잔디의 교잡종(H) 154개체의 엽폭 크기는 0.5–6.0mm으로 갯잔디 보다 범위가 큰 다양한 범위이며, 엽폭 크기가 3.001–4.0mm에서 99개체로 가장 많았으며 2.001–3.0mm과 4.001–5.0mm에서 총 51개체의 잔디들도 확인 되어, 4개체를 제외한 150개체의 엽폭 크기가 대체로 2.001–5.0mm의 범위로 나타났다(Fig. 4).
기존의 보고에서 들잔디와 갯잔디 등의 한국잔디류의 엽폭, 종자길이 및 종자 폭의 특성은 유전적 주요 형질을 반영하고 있으며 환경적으로 영향을 적게 받아 종의 분류 동정하는데 매우 유용한 형질로 판단되며, 잎의 길이(엽신 길이), 화서 길이, 화수 길이 및 화수당 종자 수 등의 특성은 한국잔디류 종 분류 동정하는데 주요한 필수 형질로 보기 어렵다고 보고하였다(Hong and Yeam, 1985; Yang et al., 1995; Choi et al., 1997; Bae et al., 2010; Lee et al., 2013). 한국잔디류는 봄 또는 여름에 종자가 맺혀 종자를 관찰하는데 시간적 제약이 있기 때문에, 항상 관찰이 가능한 엽폭 크기를 이용하여 주로 분류 동정해 왔다(Hong and Yeam, 1985; Yang et al., 1995; Choi et al., 1997; Bae et al., 2010; Lee et al., 2013; Yang et al., 2021a, 2021b).
본 연구에서는 분자생물학적으로 분류 동정된 들잔디, 갯잔디, 들잔디와 갯잔디의 교잡종의 엽폭 크기의 범위가 대체로 들잔디(Zj)의 경우 4–5mm, 갯잔디(Zs)의 경우 2–4mm, 들잔디와 갯잔디의 교잡종(H)의 경우 3–4mm로 분석되었다(Fig. 4). 그러나, 일부 들잔디의 6개체들(2–3mm)과 갯잔디 2개체들(4–5mm), 들잔디와 갯잔디의 교잡종의 51개체들(2–3mm과 4–5mm)이 존재하며, 이러한 각 종별 엽폭의 크기는 서로 겹치는 범위가 있기 때문에 외부형태학적 주요 특성인 엽폭 크기는 들잔디와 갯잔디의 교잡종을 확실히 분류 동정하기에 쉽지 않음을 확인할 수 있었다(Fig. 4). 다양한 장소에서 수집 보존중인 한국잔디류의 경우, 확연히 다른 환경인 고지대의 국립공원 산에서 수집된 들잔디나 갯벌 또는 바닷물이 인접한 바닷가 바위에서 수집된 갯잔디들은 외부형태학적인 명확한 차이 때문에 종 분류 동정은 쉽게 가능하다. 그러나, 해안가와 섬지역 같은 바다와 가까운 환경에서 수집된 들잔디, 갯잔디 및 들잔디와 갯잔디의 교잡종은 외형이 유사하여 외부형태학적 분류동정이 쉽지 않기 때문에(Yang et al., 1995; Hong et al., 2017), 신속정확한 분자생물학적 분류 동정법이 요구됨을 알 수 있었다.
본 연구에서는 기존에 본 연구팀에서 확립한 ITS 기반 CAPS 마커를 활용하여 한국 주요 지역에서 수집하여 보존 중인 다량의 한국잔디류 유전자원을 추가 분류동정 하였으며, ITS 기반 TaqMan SNP genotyping assay 분석법을 개발하여 좀 더 신속 정확하게 분자생물학적으로 다량의 한국잔디류를 분류동정 하였다. 또한 분자생물학적 분류동정된 들잔디, 갯잔디 및 교잡종의 서식환경별 분포를 조사하였으며, 주요 외부형태적 분류 특성인 엽폭을 조사하여 각 종들과 비교 분석하여 분자생물학적 분류 동정법의 적용 가능성을 제시하였다. 본 연구에서 최종 개발된 분자생물학적 분류 동정법을 이용하면 우리나라에 자생하는 들잔디, 갯잔디 및 들잔디와 갯잔디의 교잡종의 유용 형질의 유전자원을 신속하게 식별하여 유전자원 보존 및 관리, 교잡육종 및 유용 신품종 개발 시 계통분석에 활용될 수 있을 것으로 기대된다.